
C’est l’un des tests réalisé par OVALIDE (tableau 1.Q.7.CHSP)

Pour l’exemple, voici le résultat pour un RSS où 2 séjours se superposent. Ceux-ci apparaissent bien dans le tableau OVALIDE.
Il nous faut donc trouver les séjours en cause.
Cerner la question
Les fichiers à utiliser
Il s’agit de données MCO, nous allons donc avoir besoin du RSS correspondant à la période.
Nous ne devons pas utiliser le jeu produit par synthese_sejours dans l’article Les fonctions (premier contact) car un séjour peut être composé de plusieurs RUM disjoints comme par exemple si vous traitez vos séances en séjour unique avec de multiples venues.
Nous allons donc plutôt nous servir directement des RUM donc du RSS non retravaillé.
Problème, le RSS ne comporte pas d’identifiant unique du patient (depuis cette année, 2022, il y a bien l’INS dans le RSS format 121 mais celui-ci est facultatif et pas encore maitrisé dans tous les établissements).
En fait, l’identifiant unique du patient « IPP », existant obligatoirement au sein de l’établissement ou du groupement d’établissements se trouve dans le VIDHOSP entre les positions 364 et 373 et dans le même fichier, on trouve le « Numéro de séjour » (NDA chez nous, mais certains utilisent d’autres petits noms) en positions 29 à 48.
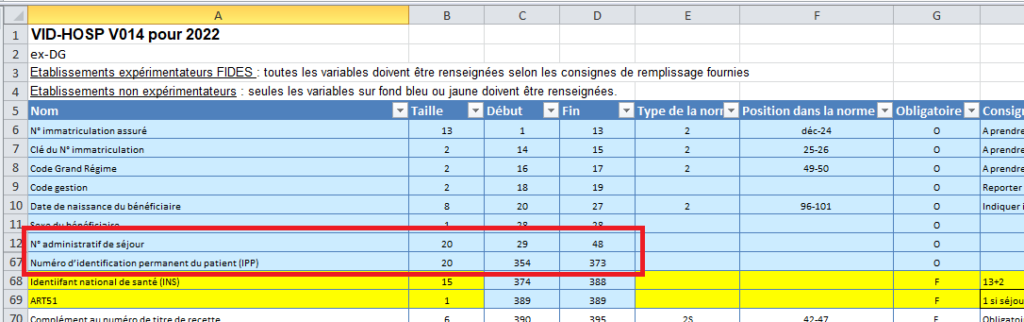
Celui-ci est réputé unique par patient (sauf en cas de doublon non réconcilié, ce qui ne devrait pas arriver).
Nous avons donc trouvé un moyen d’individualiser chaque patient bénéficiant d’un séjour.
Quel traitement appliquer pour faire ressortir les séjours qui se chevauchent ?
Pour chaque RUM nous allons chercher si sa date d’entrée intervient pendant un autre RUM du même patient.
Par exemple :

D’abord nous éliminons les croisements où S1 et S2 sont le même RUM :
(NRUM.S1 != NRUM.S2)
Le résultat d’une jointure est une produit cartésien entre les 2 jeux. S1 dans S2 est présent dans la jointure mais S2 dans S1 aussi donc il n’est pas nécessaire de tester la présence de DSORT entre DENT et DSORT de l’autre séjour.
DENT.S1 >= DENT.S2 et
DENT.S1 <= DSORT.S2
Problème, dans le cas d’un séjour multiRUM, un RUM peut enchainer un autre et alors DENT.S2 == DSORT.S1 et c’est normal ! il faut donc alors retirer DSORT.S2 du test « simple » et écrire un test spécifique pour DENT = DSORT selon si nous sommes dans le même séjour (le même NDA). Notre test devient donc :
(DENT.S1 > DENT.S2 et DENT.S1 < DSORT.S2) ou
(DENT.S1 == DSORT.S2 et NDA.S1 != NDA.S2)
En conclusion
Il nous faut lire le RSS (les champs NDA,DENT,DSORT, NRUM au minimum) et le VIDHOSP (les champs NDA et IPP au minimum).
« Désanonymiser » le RSS en le croisant avec le VIDHOSP.
Puis tester l’intersection.
Passons au code
Comme d’habitude nous commençons par charger les libraires nécessaires. Ici, juste dplyr et readr.
library(dplyr)
library(readr)
Chargement des fichiers
Comme dans les autres articles, pour illustrer les fichiers se nommeront « DEF.RSS » et « DEF.VID ».
Nous allons simplement utiliser l’équivalent de notre fonction lire_rss() que je limite, pour illustrer, uniquement aux champs strictement nécessaires.
lit_rss <- function(fichier){
read_fwf(file = fichier,
col_positions = fwf_cols(NDA = c(48, 67),
DENT = c(93, 100),
DSORT= c(103, 110),
NRUM = c(68, 77)
),
col_types = paste(rep("c", times = 4), collapse = "")
) %>%
mutate( DENT = as.Date(DENT, format = "%d%m%Y"),
DSORT = as.Date(DSORT, format = "%d%m%Y")
)
}
Puis nous écrivons la même fonction adaptée aux champs nécessaires du VIDHOSP :
lit_vidhosp <- function(fichier){
read_fwf(file = fichier,
col_positions = fwf_cols(NDA = c(29, 48),
IPP = c(364, 373)
),
col_types = paste(rep("c", times = 2), collapse = "")
)
}
Nous pouvons alors « désanonymiser » le RSS en mettant en place une jointure :
rss <- lit_rss("DEF.RSS")
vidhosp <- lit_vidhosp("DEF.VID")
rssd <- rss %>% left_join(vidhosp %>% unique, by = "NDA")
Dans le cas présent, j’ai précisé le by= "NDA" mais sachez qu’il n’est pas obligatoire car non ambigu entre les deux jeux de données. Je vous conseille de le conserver vous aussi car cela aide à relire et comprendre a posteriori le code.
A noter le %>% unique qui s’assure que les couples NDA/IPP sont uniques dans le jeu de données.
Pour la petite histoire, l’un des logiciels de GAP que nous utilisons nous a sorti il y a quelques mois des NDA pointant vers plusieurs IPP, cela bien entendu casse la fonctionnalité si on se limite à un croisement par NDA car avec les éléments disponibles il faudrait faire un choix à l’aveugle du bon couple NDA/IPP. La bonne réponse à ce problème est bien sûr de réparer le VIDHOSP et non de contourner dans le code de traitement…
Pourquoi un left_join et pas un inner_join ?
La différence principale est que si on met un inner_join(), l’absence de donnée dans la table VIDHOSP (une extraction qui ne serait pas synchronisée) supprimerait les RUM correspondant du RSS résultant. Tandis qu’un left_join() garde toutes les lignes de la table « de gauche » et met des valeurs NA sur les valeurs absentes de la table de droite.
Dans notre recette, nous n’attendons pas de faire d’autres traitements dessus mais si c’était le cas, notre RSS ne serait plus exhaustif…
Croisons le jeu de données sur lui-même
> rss %>% inner_join(rss, by = "IPP", suffix = c(".S1",".S2")) %>%
filter(NRUM.S1 != NRUM.S2 &
(DENT.S1 > DENT.S2 & DENT.S1 < DSORT.S2) |
(DENT.S1 == DSORT.S2 & NDA.S1 != NDA.S2)
) %>%
select(NDA.S1, NDA.S2, DENT.S1, DENT.S2, DSORT.S2)
# A tibble: 2 x 5
NDA.S1 NDA.S2 DENT.S1 DENT.S2 DSORT.S2
<chr> <chr> <date> <date> <date>
1 422016601 422021751 2022-03-30 2022-03-27 2022-04-01
2 422016601 422021751 2022-03-28 2022-03-27 2022-04-01
Et voilà, nous avons trouvé nos 2 erreurs. (Dans la pratique, il s’agissait de séances de dialyses créées à tort au cours d’un séjour hospitalier)
Conclusion
Bravo, vous avez désormais un exemple pour trouver des séjours simultanés. Et pourquoi ne pas garder cette possibilité dans un petit coin en en faisant une/des fonctions que vous pourrez réutiliser (j’ai rajouté des paramètres dans le jeu en sortie, libre à chacun de les enlever par un select() de plus) ?
desanonymise <- function(jeu,vidhosp) {
jeu %>% left_join(vidhosp %>% select(NDA,IPP),by = "NDA")
}
sejours_simultanes <- function(rssd) {
rssd %>% inner_join(rssd, by = "IPP", suffix = c(".S1",".S2")) %>%
filter(NRUM.S1 != NRUM.S2 &
(DENT.S1 > DENT.S2 & DENT.S1 < DSORT.S2) |
(DENT.S1 == DSORT.S2 & NDA.S1 != NDA.S2)
) %>%
select(NDA.S1, NRUM.S1,
NDA.S2, NRUM.S2,
DENT.S1, DSORT.S1,
DENT.S2, DSORT.S2,
IPP)
}
Et cela s'appelle en faisant :
> sejours_simultanes(desanonymise(rss,vidhosp))
ou avec le pipe à la magrittr :
> rss %>% desanonymise(vidhosp) %>% sejours_simultanes

