
La Réhabilitation Accélérée Après Chirurgie (RAAC) est une évolution des pratiques visant à raccourcir le délai de retour à domicile après une chirurgie lourde. Cela aboutit théoriquement à une réduction de la DMS. Il s’agit donc d’un bon indicateur de fonctionnement de ce changement de pratique.
Si la diminution de la durée de séjour est aussi à l’intérêt de l’établissement, l’inconvénient potentiel serait en terme de valorisation.
Les séjours « lourds » de patients présentant des comorbidités générant sévérité, pourraient être tellement raccourcis que la durée de séjour impactera le déclenchement des GHM à sévérité. Ainsi un séjour « traditionnel » de PTH de 5 jours qui devrait finir en GHM 08C483 (donc sévérité 3), si la durée de séjour passe à 2 jours il ne groupera plus qu’en niveau 1 08C481. La différence de valorisation (en ex-DG) est alors 170 1,73 €, passant de 6 821,69 € à 5 119,96 €.
Lors de la définition de la mesure RAAC, il a donc été fixé qu’il n’y a plus de borne de déclenchement de sévérité sur les séjours étiquetés RAAC permettant donc d’optimiser la DS de ces séjours sans impacter négativement la rentabilité.
La problématique
La problématique que nous allons explorer dans cet article est « comment l’exploration du PMSI en R nous permettra de suivre les répercussions de la filière RAAC de l’établissement ? ».
Il y a cependant là un « problème » : un séjour RAAC doit grouper dans le GHM correspondant à sa durée de séjour avec les règles habituelles de borne basse. Ainsi dans le RSS, le GHM porté dans l’exemple ci-dessus sera toujours 08C481 même s’il s’agit d’un séjour avec des CMA 3. Pourtant la valorisation ne se fera pas dans le GHS 2885 (SEV=1) mais 2887 (SEV=3). Or le GHS n’est pas disponible dans le RSS car calculé du côté du « payeur » (par GENRSA).
Approche en R
Configuration initiale
Pour la lisibilité et la praticité nous allons utiliser les librairies dplyr et tidyr, toutes deux membres du tidyverse.
library(dplyr)
library(tidyr)
Pour sélectionner certaines valeurs pertinentes, nous allons spécifier une variable qui contiendra notre statut. Dans mon cas, étant dans un ESPIC, nous sommes assimilés public :
statut = "public" # mais on mettrait "prive" si Ex-OQN
La source des données séjours : le RSA
Le RSA est le fichier de sortie anonymisé de GENRSA (format 2022, version 227 à la date de rédaction de l’article), pendant du RSS en entrée non anonymisé.
Dans le RSA, nous disposons de plusieurs champs plus pertinents pour notre analyse :
- le GHM produit est disponible à l’adresse 31-36 (pour le GHM en entrée de GENRSA, que j’appellerai
GHMIN) et 42-47 (pour le GHM en sortie de GENRSA, respectivementGHMOUT). En théorie, le GHMIN - le
GHSde valorisation est à l’adresse 96-99 - la durée de séjour est à 71-74 (qu’on appellera
DS) - et enfin le marqueur
RAACà 199-199
Le fichier en lui-même se trouve dans l’archive « out.zip » générée par GENRSA. Le but n’étant pas d’expliquer comment on dézippe une archive, je vous laisse le faire à la main et placer le fichier à l’emplacement de votre choix. Fixons ensuite cet emplacement dans une variable pour pouvoir le retrouver et changer rapidement :
# fichierRSA <- "chemin complet vers le fichier RSA"
# par exemple, dans le cadre de cet article, chez moi :
fichierRSA <-"~/R/M12.rsa"
De la même façon que nous avons déjà importé des éléments du fichier des UM et du RSS, nous allons faire de même avec le RSA. De plus nous allons en profiter pour isoler dès maintenant la racine et la sévérité liées au GHM (extraire les 5 premiers puis dernier caractères du GHM complet) :
RSA <- read_fwf( file = fichierRSA,
col_positions = fwf_cols(GHMIN = c(31, 36),
RGHMIN = c(31, 35),
SEVIN = c(36, 36),
GHMOUT = c(42, 47),
RGHMOUT= c(42, 46),
SEVOUT = c(47,47),
GHS = c(96, 99),
DS = c(71, 74),
RAAC = c(199, 199)
),
col_types = "cccccccic"
)
En théorie GHMIN et GHMOUT devraient être égaux sinon c’est que vous avez une erreur de groupage dans le logiciel qui gère le PMSI.
Pour la beauté du traitement, vous pouvez trouver ces séjours (anonymisés) en faisant :
RSA %>% filter(GHMIN != GHMOUT)
(mais cela n’a rien à voir avec notre problème. 😉)
Si d’autres éléments du RSA vous intéressent, le descriptif du format est disponible en annexe du manuel de GENRSA récupérable sur le site de l’ATIH, rubrique « Téléchargement ».
Les données complémentaires
Pour ne pas alourdir l’article, nous allons utiliser principalement les données de la librairie refpmsi. Il faut donc l’avoir installée telle que décrit dans l’article en lien.
library(refpmsi)
si vous voulez, vous pouvez probablement utiliser la librairie nomensland cependant elle est moins à jour, il vous faudra aussi adapter le code… Vous pouvez aussi utiliser directement des tables personnelles mais je vous laisse alors gérer leur chargement.
Les racines de GHM RAAC
La RAAC n’est instaurable que dans une partie limitée de l’arbre des GHMs, à ce jour :
- 04C02 : Interventions majeures sur le thorax
- 04C04 : Interventions sous thoracoscopie
- 06C03 : Résections rectales
- 06C04 : Interventions majeures sur l’intestin grêle et le côlon
- 06C07 : Interventions mineures sur l’intestin grêle et le côlon
- 06C16 : Interventions sur l’oesophage, l’estomac et le duodénum pour tumeurs malignes, âge supérieur à 17 ans
- 07C09 : Interventions sur le foie, le pancréas et les veines porte ou cave pour tumeurs malignes
- 08C22 : Interventions pour reprise de prothèses articulaires
- 08C24 : Prothèses de genou
- 08C25 : Prothèses d’épaule
- 08C27 : Autres interventions sur le rachis
- 08C48 : Prothèses de hanche pour des affections autres que des traumatismes récents
- 08C52 : Autres interventions majeures sur le rachis
- 10C13 : Interventions digestives autres que les gastroplasties, pour obésité
- 11C02 : Interventions sur les reins et les uretères et chirurgie majeure de la vessie pour une affection tumorale
- 12C11 : Interventions pelviennes majeures chez l’homme pour tumeurs malignes
- 13C03 : Hystérectomies
- 13C14 : Exentérations pelviennes, hystérectomies élargies ou vulvectomies pour tumeurs malignes
- 13C15 : Exentérations pelviennes, hystérectomies élargies ou vulvectomies pour affections non malignes
Il nous faut donc un vecteur de type caractère reprenant ces valeurs. Cependant cette liste n’est pas disponible dans refpmsi, il nous faut donc la créer :
GHMRAAC <- c("04C02", "04C04",
"06C03", "06C04","06C07","06C16",
"07C09",
"08C22", "08C24", "08C25", "08C27", "08C48", "08C52",
"10C13", "11C02",
"12C11",
"13C03", "13C14", "13C15")
Les données de GHM
Il nous faut les données de regroupement GHM afin d’avoir des jolis libellés. Nous n’avons besoin que de ça donc limitons les colonnes inutiles.
RGHMs <- refpmsi("rghm_regroupement", 2022) %>%
select(rghm, rghm_lib)
RGHMs
# A tibble: 668 x 2
rghm rghm_lib
<chr> <chr>
1 01C03 Craniotomies pour traumatisme, âge supérieur à 17 ans
2 01C04 Craniotomies en dehors de tout traumatisme, âge supérieur à 17 ans
3 01C05 Interventions sur le rachis et la moelle pour des affections neurologiques
4 01C06 Interventions sur le système vasculaire précérébral
5 01C08 Interventions sur les nerfs crâniens ou périphériques et autres interventions sur le système nerveux
6 01C09 Pose d'un stimulateur cérébral
7 01C10 Pose d'un stimulateur médullaire
8 01C11 Craniotomies pour tumeurs, âge inférieur à 18 ans
9 01C12 Craniotomies pour affections non tumorales, âge inférieur à 18 ans
10 01C14 Libérations de nerfs superficiels à l'exception du médian au canal carpien
# ... with 658 more rows
# i Use `print(n = ...)` to see more rows
Exercice :On peut filtrer pour ne garder que ceux qui correspondent aux racines RAAC. Je vous laisse chercher comment faire.
Les données de GHM, GHS et valorisation
Elles se trouvent en 2 versions : public (Ex-DG) et privé (Ex-OQN), nous allons utiliser la variable que nous avons définie plus haut pour choisir le bon :
GHSs <- refpmsi(!!paste("ghs_", statut, sep=""), 2022)
paste() est la fonction permettant de coller des chaines de caractères l’une à la suite de l’autre, séparées par la valeur de sep (ici la chaine vide).
Vous vous demandez probablement pourquoi le !! (on appelle ça un « bang-bang« ). C’est une fonction qui force l’exécution de la fonction qu’il précède. refpmsi est programmé pour que son 1er paramètre, referentiel, soit accepté avec ou sans guillemets (ainsi refpmsi("ghs_public") est équivalent à refpmsi(ghs_public)). Sans le bang-bang, refpmsi cherche une table dont le nom est un appel de la fonction paste, et non dont le nom est le résultat de la fonction. Vous vous doutez bien qu’un tableau ne peut pas être initialisé avec un appel de fonction qui par définition est une valeur interne, variable et fugace.
Quoi qu’il en soit, nous récupérons bien notre table :
GHSs
# A tibble: 5,565 x 10
ghs ghm ghm_lib ghm_bb ghm_bh ghs_t~1 ghs_exb ghs_exh annee~2 ghs_i~3
<chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <chr> <chr>
1 22 01C031 Craniotomies pour traumatisme, âge supérieur à 17~ 0 11 3742. 0 111. 2022 NA
2 5071 01C031 Craniotomies pour traumatisme, âge supérieur à 17~ 0 0 2806. 0 0 2022 NA
3 23 01C032 Craniotomies pour traumatisme, âge supérieur à 17~ 0 19 6644. 0 91.4 2022 NA
4 5071 01C032 Craniotomies pour traumatisme, âge supérieur à 17~ 0 0 2806. 0 0 2022 NA
5 24 01C033 Craniotomies pour traumatisme, âge supérieur à 17~ 0 64 11989. 0 71.0 2022 NA
6 5071 01C033 Craniotomies pour traumatisme, âge supérieur à 17~ 0 0 2806. 0 0 2022 NA
7 25 01C034 Craniotomies pour traumatisme, âge supérieur à 17~ 12 124 16246. 405. 301. 2022 NA
8 5071 01C034 Craniotomies pour traumatisme, âge supérieur à 17~ 0 0 2806. 0 0 2022 NA
9 26 01C041 Craniotomies en dehors de tout traumatisme, âge s~ 0 12 5866. 0 136. 2022 NA
10 65 01C041 Craniotomies en dehors de tout traumatisme, âge s~ 0 12 12962. 0 136. 2022 NA
# ... with 5,555 more rows, and abbreviated variable names 1: ghs_tarif, 2: annee_pmsi, 3: ghs_intermediaire
# i Use `print(n = ...)` to see more rows
(Vous pouvez voir au passage dans la structure du tableau qu’il existe un lien entre ghs et ghm, mais attention, il ne s’agit pas toujours d’une relation un-à-un, comme vous le savez déjà). Il se trouve que cela est causé par les GHM/GHS UHCD (tous ces GHM d’une même racine pointent vers le même GHS). La liste des GHM UHCD existant dans refpmsi, nous pouvons les supprimer (la RAAC ne peut pas découler d’un séjour en urgence) par :
GHSs <- GHSs %>% anti_join(refpmsi(!!paste("ghm_monorum_uhcd_", statut, sep=""), 2022), by = "ghs")
un anti_join() est une jointure « filtrante » définie par dplyr. Elle supprime du jeu source, toutes les lignes se trouvant dans le jeu secondaire en se basant sur le pivot renseigné par by=. Le code ci-dessus permet donc de retirer de GHSs les lignes concernant des GHS présents dans le réferentiel « monorum UHCD » (la liste n’est pas genrée public/privé dans cette utilisation, uniquement si on veut valoriser de l’UHCD, mais c’était l’occasion de remettre le paste() vu plus haut).
Enfin pour être tranquille, nous filtrons uniquement les lignes correspondant aux racines RAAC car alors il n’y aura plus de doublons :
GHSs <- GHSs %>% filter(substr(ghm, 1, 5) %in% GHMRAAC)
Bien sûr nous pouvons, grace à des pipes %>% écrire tout cela d’une traite vu que nous n’avons pas besoin des étapes intermédiaires :
GHSs <- refpmsi(!!paste("ghs_", statut, sep=""), 2022) %>%
anti_join(refpmsi(!!paste("ghm_monorum_uhcd_", statut, sep=""), 2022), by = "ghs") %>%
filter(substr(ghm, 1, 5) %in% GHMRAAC)
(Et vous avez ainsi la piste pour la solution à l’exercice ci-dessus)
L’interrogation
Dans la suite de l’article, je vous propose de d’abord essayer par vous même de répondre à la question avant de lire le contenu.
J’utilise un jeu réduit (pour que vous puissiez vérifier à la main si besoin) :
> RSA
A tibble: 12 x 9
GHMIN RGHMIN SEVIN GHMOUT RGHMOUT SEVOUT GHS DS RAAC
<chr> <chr> <chr> <chr> <chr> <chr> <chr> <int> <int>
1 04C021 04C02 1 04C021 04C02 1 1005 5 1
2 04C021 04C02 1 04C021 04C02 1 1005 0 0
3 04C023 04C02 3 04C023 04C02 3 1007 6 0
4 04C021 04C02 1 04C021 04C02 1 1005 1 1
5 04C022 04C02 2 04C022 04C02 2 1006 6 0
6 04C021 04C02 1 04C021 04C02 1 1007 1 1
7 04C024 04C02 4 04C024 04C02 4 1008 6 0
8 04C022 04C02 2 04C022 04C02 2 1006 6 1
9 04C041 04C04 1 04C041 04C04 1 1013 5 1
10 04C041 04C04 1 04C041 04C04 1 1013 0 1
11 04C041 04C04 1 04C041 04C04 1 1013 8 0
12 06C034 06C03 4 06C034 06C03 4 1938 8 0
1ère question posée : « Les effectifs de la RAAC par racine de GHM »
Cette question est simple, nous cherchons les séjours ayant une valeur de champ RAAC différente de « 0 ». En théorie le champ ne peut pas être vide ; dans le doute, on pourrait rajouter dans le filter & !is.na(RAAC) & RAAC !="")si cela pouvait arriver.
RSA %>% filter(RAAC != "0")
Cette ligne va retourner une table avec tous les séjours RAAC. Mais nous cherchons à calculer des effectifs. Nous devons donc regrouper les lignes pour calculer les effectifs respectifs de chaque RGHM et selon l’indicateur RAAC. Nous allons utiliser group_by/summarise pour cela :
RSA %>% group_by(RGHMOUT,RAAC) %>%
summarise(n=n())
Il faut indiquer dans group_by() les champs devant être conservés et sur lesquels se feront le regroupement, ici le rghm et l’indicateur RAAC puis dans summarise() les fonctions de regroupement : ici n() qui compte les éléments dans chaque groupe.
Nous allons obtenir une sortie de ce genre (un RSA d’uniquement 12 lignes…)
`summarise()` has grouped output by 'RGHMOUT'. You can override using the `.groups` argument.
# A tibble: 5 x 3
# Groups: RGHMOUT [3]
RGHMOUT RAAC n
<chr> <int> <int>
1 04C02 0 4
2 04C02 1 4
3 04C04 0 1
4 04C04 1 2
5 06C03 0 1
Les données y sont mais pas très lisibles s’il y en avait plus. Nous pouvons alors utiliser une fonction de tidyr pour les présenter plus proprement à la manière des tableaux dynamiques d’Excel, pivot_wider(). Grace à la puissance du pipe %>%, nous n’avons qu’à la rajouter à la fin de l’étape précédente, avec un petit mutate() de propreté :
RSA %>% group_by(RGHMOUT,RAAC) %>%
summarise(n=n()) %>%
mutate(RAAC = ifelse(RAAC == 0, "NonRAAC", "RAAC")) %>%
pivot_wider(names_from = RAAC, values_from =n, values_fill =0)
`summarise()` has grouped output by 'RGHMOUT'. You can override using the `.groups` argument.
# A tibble: 3 x 3
# Groups: RGHMOUT [3]
RGHMOUT Non RAAC RAAC
<chr> <int> <int>
1 04C02 4 4
2 04C04 1 2
3 06C03 1 0
2ème question : « Quelle est la DMS des séjours selon leur statut RAAC ou non RAAC ? »
Nous avons un champ dans notre RSA contenant cette donnée, nous l’avons appelé DS.
Il nous suffit donc de modifier le calcul dans le regroupement. Une moyenne s’établit avec la fonction mean() auquel on passe le vecteur de données à moyenner.
RSA %>% group_by(RGHMOUT,RAAC) %>%
summarise(DMS=mean(DS)) %>%
mutate(RAAC = ifelse(RAAC == 0, "NonRAAC", "RAAC")) %>%
pivot_wider(names_from = RAAC, values_from =DMS, values_fill =NA)
RGHMOUT NonRAAC RAAC
<chr> <dbl> <dbl>
1 04C02 4.5 3.25
2 04C04 8 2.5
3 06C03 8 NA
Le values_fill = NA n’est pas obligatoire car c’est la valeur par défaut. NA permet dans le cas présent de différencier une DMS à 0 et un recueil absent (dans le jeu de base, il n’y a pas de RAAC en 06C03)
3ème question : « Combien de séjours bénéficient du débornage des sévérités ? »
Nous allons utiliser la table GHSs que vous avons récupérée précédemment pour cela.
En liant les deux tables RSA et GHSs par leurs champs GHS/ghs respéctifs, nous pouvons trouver le GHM qui a été utilisé pour calculer le GHS :
RSA %>% inner_join(GHSs %>% select(ghs, ghm), by = c("GHS" = "ghs"))
# A tibble: 12 x 8
GHMIN SEVIN GHMOUT SEVOUT GHS DS RAAC ghm
<chr> <chr> <chr> <chr> <chr> <int> <int> <chr>
1 04C021 1 04C021 1 1005 5 1 04C021
2 04C021 1 04C021 1 1005 0 0 04C021
3 04C023 3 04C023 3 1007 6 0 04C023
4 04C021 1 04C021 1 1005 1 1 04C021
5 04C022 2 04C022 2 1006 6 0 04C022
6 04C021 1 04C021 1 1007 1 1 04C023
7 04C024 4 04C024 4 1008 6 0 04C024
8 04C022 2 04C022 2 1006 6 1 04C022
9 04C041 1 04C041 1 1013 5 1 04C041
10 04C041 1 04C041 1 1013 0 1 04C041
11 04C041 1 04C041 1 1013 8 0 04C041
12 06C034 4 06C034 4 1938 8 0 06C034
Et de là nous pouvons extraire le niveau de sévérité « SEVVALO » appliqué pour définir réellement le GHS et la valorisation.
(...) %>% mutate(SEVVALO = substr(ghm,6,6))
A nouveau pour des raison de praticité, un petit coup de mutate(), group_by/summarise() et pivot_wider()
(...) %>% mutate(LIB = ifelse(RAAC == 0,
paste("NR:", SEVVALO, sep = ""),
paste("R:", SEVOUT,"->",SEVVALO,sep="")
)
) %>%
group_by(RGHMOUT, LIB) %>% summarise(n = n()) %>%
pivot_wider(names_from = LIB, values_from = n, values_fill = 0)
# A tibble: 3 x 8
# Groups: RGHMOUT [3]
RGHMOUT `NR:1` `NR:2` `NR:3` `NR:4` `R:1->1` `R:1->3` `R:2->2`
<chr> <int> <int> <int> <int> <int> <int> <int>
1 04C02 1 1 1 1 2 1 1
2 04C04 1 0 0 0 2 0 0
3 06C03 0 0 0 1 0 0 0
Ce qui nous permet de voir que sur le jeu d’exemple, 1 séjour a été valorisé en niveau 3 grâce à la RAAC alors qu’il aurait dû être sans sévérité sinon, 4 séjours RAAC étaient sans sévérité de base et le sont restés et 1 séjour était de sévérité 2 et l’est resté.
Améliorer encore la présentation
En début d’article nous avons récupéré les libellés des GHM, autant les utiliser. Tout le monde ne maitrise pas la lecture des codes GHM (en fait, personne à part nous…). Alors il est possible d’améliorer la présentation en rajoutant ce fameux libellé :
(...) %>% left_join(RGHMs %>% select(rghm,rghm_lib),by = c("RGHMOUT" = "rghm"))
`summarise()` has grouped output by 'RGHMOUT'. You can override using the `.groups` argument.
# A tibble: 3 x 9
# Groups: RGHMOUT [3]
RGHMOUT `NR:1` `NR:2` `NR:3` `NR:4` `R:1->1` `R:1->3` `R:2->2` rghm_lib
<chr> <int> <int> <int> <int> <int> <int> <int> <chr>
1 04C02 1 1 1 1 2 1 1 Interventions majeures sur le thorax
2 04C04 1 0 0 0 2 0 0 Interventions sous thoracoscopie
3 06C03 0 0 0 1 0 0 0 Résections rectales
et pour mettre la colonne au bon endroit, vous pouvez utiliser relocate() :
(...) %>% relocate(LIBELLE=rghm_lib,.after=RGHMOUT)
#Notez que je profite pour renommer la colonne par la même occasion
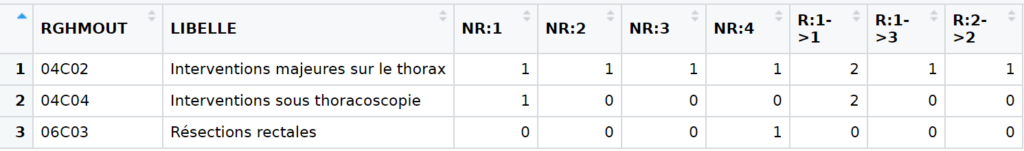
Et voilà le résultat final dans l’afficheur de RStudio. 👌
Conclusion (temporaire)
Nous allons nous arrêter là pour le moment, je vous laisse expérimenter avec vos propres données et vous poser d’autres questions en rapport avec la RAAC (ou d’autres champs du RSA). Si vous vous retrouvez coincés, n’hésitez pas à utiliser les commentaires ci-dessous et je vous accompagnerai.
La prochaine fois, nous allons écrire une fonction de valorisation de séjour générique. Nous pourrons alors poser des questions telles que « La RAAC rapporte-t-elle de l’argent à l’établissement ? », calculer des PMCT et d’autres indicateurs et vous pourrez ainsi accompagner la prise de décision de la direction et les praticiens sur l’efficience de leurs parcours RAAC.

Bonjour Fred,
à la place du summarise(n = n()) , tu peux utiliser tally() que j’ai adopté il y a peu car plus rapide a taper.
@+
Bonjour Blaise,
On peut aussi utiliser
count()qui est encore plus rapide car regroupant legroup_by()et lesummarise():RSA %>% count(RGHMOUT,RAAC)donne exactement le même résultat.(Le but était d’introduire le couple polyvalent group_by() %>% summarise(), il est assez rare de faire isolément uniquement le décompte.)
Extrêmement intéressant et didactique.
Je commence seulement à m’interesser à R et je trouve la un exercice pratique vraiment génial.
Bravo à vous d’avoir mis à la disposition des membres de cnim.io ce lien.
Rémi Bonnaire
DIM Territoire GHT Plaine de France