Mars-avril c’est cette période de l’année particulière où le monde médico-économique hospitalier retient son souffle en attendant la publication des arrêtés.
Je vais en profiter pour parler d’une phase impérative lors de la réalisation d’une analyse, c’est la phase de prétraitement des données. Et ce sera l’occasion de se pencher sur l’utilisation de « feuilles de calcul » au format EXCEL en entrée.
(Dans la suite de l’article, je ne vais parler que de l’arrêté MCO-HAD (qui contient aussi un peu d’externe) pour ne pas trop faire trop long, mais le principe est le même pour chaque champ.)
Que sont les « arrêtés » ?
Pour ceux qui sont frais dans la profession de DIM ou qui ne connaissent pas (encore) le fonctionnement, le 1er mars est le jour (pour le moment) où les tarifs hospitaliers changent. Pour cela, après des arbitrages en hauts lieux, sont publiés deux documents principaux qui vont s’imposer aux établissements :
- L’arrêté prestation qui définit les prestations facturables par les établissements de santé
- et l’arrêté tarifaire qui définit les tarifs respectifs de ces prestations pour les établissements selon s’ils sont privés à but lucratif (ex-OQN) ou bien public/privés à but non lucratif (ex-DG)
Ces arrêtés contiennent du jargon technique législatif mais sont surtout accompagnées d’annexes qui sont produites actuellement systématiquement en retard de plusieurs semaines d’où la fébrilité des directions financières car entre le 1er mars et la date de publication de ces données, on ne sait pas comment sera valorisée l’activité réalisée (et il est donc impossible aussi de faire du prospectif).
Une fois ces documents nationaux produits, chaque ARS a pour mission de transmettre officiellement un arrêté à chaque établissement. On le reçoit principalement sous forme d’un PDF, réglementaire mais totalement inutilisable informatiquement. Mais à partir de la réception, on peut reprendre la facturation. C’est du réglementaire quoi…
La période des arrêtés est aussi celle de changement éventuelle des règles de groupage, donc de la fonction groupage et des formats des fichiers d’export et d’envoi. En cette année 2025, il n’y a pas de modification profonde.
Pour ceux qui désirent lire les arrêtés, il se trouvent sur Légifrance. Pour 2025, à : https://www.legifrance.gouv.fr/jorf/id/JORFTEXT000051439149.
Les différents articles, tableaux et annexes qui font alors loi sont ensuite repris dans des fichiers au format « Excel » produits par l’ATIH et mis à disposition sur une page annuelle dédiée. A nouveau, pour 2025 : https://www.atih.sante.fr/arretes-prestations-et-tarifaires-mco-had-2025. Bien sûr, il existe la même chose pour les autres champs PMSI.
Lorsqu’on traite du PMSI, le contenu de ces documents réglementaires sont capitaux mais sont du texte (html ou PDF du JO). De par sa position d’interlocuteur principal avec les établissements (et le format facile à manier des fichiers), l’ATIH est considérée une source primaire pour les fichiers informatiquement traitables. Une autre source primaire seraient les fichiers NX de l’assurance maladie, cependant leur traitement est bien plus compliqué pour un besoin ponctuel et surtout le format NX n’est pas adapté à un prétraitement par R. Un jour peut-être les aborderai-je ici (dans une petite disgression python par exemple) mais pour aujourd’hui priorité à l’action !
Aujourd’hui nous allons nous occuper de l’arrêté PRESTATIONS, c’est à dire celui qui est disponible le plus tôt et qui ne contient pas les données de valorisation des GHS. Nous regarderons l’arrêté TARIFAIRE une prochaine fois.
Le concept de prétraitement
Il s’agit de prendre des données brutes, de les manipuler, recouper pour soit les sauvegarder dans un nouveau fichier, soit les utiliser directement.
Si nous les sauvegardons après le prétraitement, nous créons une source secondaire. C’est à dire une interprétation des données primaires à notre format.
Quand de nombreuses sources existent ou qu’il y a beaucoup de prétraitements à réaliser pour obtenir des fichiers cohérents, il est parfois intéressant de mettre en place un gestionnaire ETL. Mais même sans, le principe ETL reste pertinent.
Qu’est-ce que l’ETL ?
ETL est l’acronyme de « Extract – Transform – Load » que l’on peut traduire par « Extrait – Transforme – Sauvegarde ». Voici ci-dessous un exemple fictif d’une telle chaine :
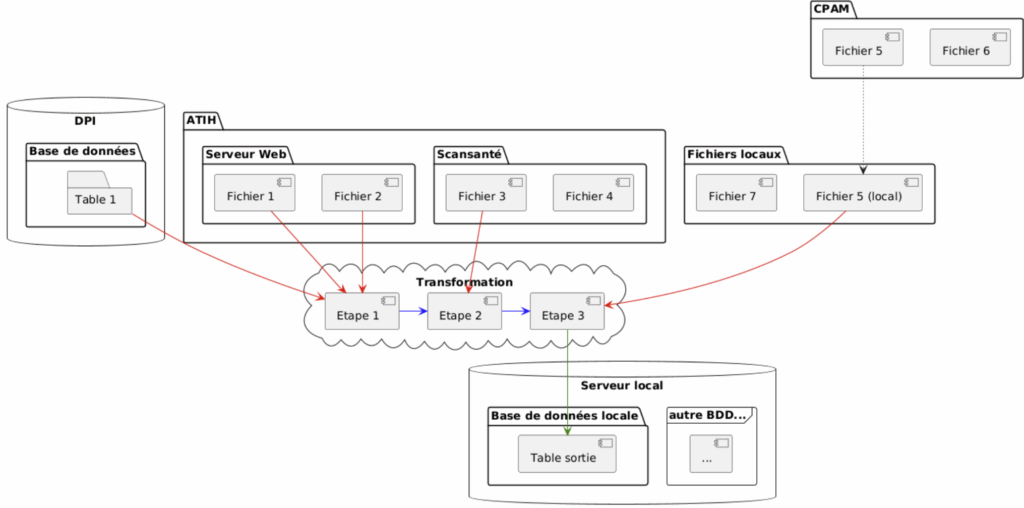
En rouge, vous avez l’extraction qui met les données brutes à disposition de la chaine de transformation (en bleu) et ensuite la finalisation par la phase de sauvegarde en vert. Une fois la sauvegarde effectuée, rien n’empêche d’utiliser cette source secondaire en entrée pour un nouveau traitement ETL.
Dans le schéma ci-dessus, le DPI peut être interrogé via une requête SQL sur la base de données. Les fichiers 1 et 2 sont récupérés directement depuis le site de l’ATIH, le fichier 3 depuis ScanSanté. Tandis que le fichier 5 est d’abord téléchargé manuellement sur notre disque dur puis utilisé. Enfin les fichiers 4, 6 et 7 ainsi que la base de données « autre BDD » existent mais ne sont pas utilisés.
R est capable de faire tout ça soit nativement soit avec quelques librairies (pour l’accès aux bases de données et bien sûr la partie traitement).
Dans la suite de l’article nous allons voir une décomposition pleins de petites chaines de ce gerne pour l’arrêté « Prestations ».
L’arrêté « Prestations »
Le fichier XLS de cet arrêté pour 2025 est récupérable à https://www.atih.sante.fr/sites/default/files/public/content/4971/arrete_prestations_annexes_2025_vdef.xlsx si vous voulez le consulter vous-même, cependant nous allons utiliser directement la capacité de R d’aller chercher des fichiers distants. S’agissant d’un fichier au format XLSX, il nous faudra aussi la librairie readxl qui permet de le manipuler.
Malheureusement, readxl ne supporte pas le chargement direct depuis une source HTTP il n’est donc pas possible d’écrire read_excel("http://serveur.example/fichier.xlsx"). Vu que nous allons y accéder plusieurs fois, il vaut mieux le sauvegarder dans un fichier temporaire que nous allons d’abord créer via tempfile().
# On charge la librairie de manipulation des fichiers excel
library(readxl)
# on stocke la source dans une chaine de caractères pour la lisibilité future
f_source <- "https://www.atih.sante.fr/sites/default/files/public/content/4971/arrete_prestations_annexes_2025_vdef.xlsx"
# on "crée" un nom de fichier temporaire et on stocke son nom
# (le fichier sera supprimé automatiquement à la fin de la session R)
f_temp <- tempfile(fileext= "xlsx")
# on télécharge le fichier
# mode = "wb" signifie de sauvegarder en mode binaire
# c'est cette commande qui crée réellement le fichier local
download.file(f_source, f_temp, mode = "wb")
# A partir d'içi nous pouvons accéder au contenu de f_temp
# par exemple lister les feuilles contenues dedans :
excel_sheets(path = f_temp)L’exécution du script ci-dessus nous donne :
(...)
> excel_sheets(path = f_temp)
[1] "Annexe 1" "Annexe 1 bis"
[3] "Annexe 1 ter" "Annexe 2"
[5] "Annexe 3 - liste 1" "Annexe 3 - liste 2"
[7] "Annexe 4 - liste 1" "Annexe 4 - liste 2"
[9] "Annexe 5 - liste 1" "Annexe 5 - liste 2"
[11] "Annexe 6" "Annexe 7 - liste 1"
[13] "Annexe 7 - liste 2" "Annexe 7 - liste 3"
[15] "Annexe 8 - liste 1" "Annexe 8 - liste 2"
[17] "Annexe 8 - liste 3" "Annexe 9 - liste 1"
[19] "Annexe 9 - liste 2" "Annexe 10"
[21] "Annexe 11 - liste 1" "Annexe 11 - liste 2"
[23] "Annexe 11 - liste 3" "Annexe 11 - liste 4"
[25] "Annexe 11 - liste 5" "Annexe 11 - liste 6"
[27] "Annexe 11 - liste 7" "Annexe 12 - liste 1"
[29] "Annexe 12 - liste 2" "Annexe 13"
[31] "Annexe 14" "Annexe 15"
[33] "Annexe 16" "Annexe 17"
[35] "Annexe 18" "Annexe 19 - liste 1"
[37] "Annexe 19 - liste 2"Il n’y a pas à dire il y a du pain sur la planche !
excel_sheets() renvoie la liste des noms des feuilles disponibles dans le fichier pointé par path=. Dans notre cas, il s’agit du numéro de l’annexe de l’arrêté, complété du numéro de liste lorsqu’il y en a plusieurs… Pas très parlant. Pour trouver l’explication du contenu, il n’y a pas d’autre possibilité que de lire le texte de l’arrêté.
Pour abréger vos souffrances, voici l’équivalence. Le nom entre [] est la façon dont je les appellerai dans la suite de l’article (la première lettre définit le type de liste A = Actes, G = GHS ou GHM, D = Diagnostics, et les autres…).
Liste des annexes
Annexe 1. Liste des forfaits dénommés “ Groupes homogènes de séjour ”. [GHS]
Annexe 1 bis. Liste des forfaits dénommés “ Groupes homogènes de séjour intermédiaires ”. [GHSI]
Annexe 1 ter. Liste des forfaits dénommés “ Groupes homogènes de séjour monorum UHCD ”. [GHSU]
Annexe 2. Liste des forfaits de l’insuffisance rénale chronique à domicile, en unité de dialyse médicalisée ou en autodialyse. [FDIAL]
Annexe 3. Listes des forfaits dénommés “ Prélèvements d’organes ” :
– liste 1. Forfaits correspondant aux prestations de séjours et de soins délivrées par l’établissement au sein duquel a été réalisé le prélèvement d’un ou plusieurs organes [POE]
– liste 2. Forfaits correspondant aux prestations de séjours et de soins délivrées par le chirurgien qui effectue l’acte de prélèvement. [POP]
Annexe 4 :
– liste 1. Actes permettant la valorisation d’une séance de radiothérapie en sus du GHS couvrant les prestations de séjour et de soins délivrées au patient [ARAD1]
– liste 2. GHS excluant le paiement des actes de radiothérapie en sus. [GRAD0]
Annexe 5 :
– liste 1. Actes permettant la facturation d’une séance de dialyse en hospitalisation en sus d’un GHS [ADIAL1]
– liste 2. Liste des GHS excluant le paiement des actes de dialyse en sus. [GDIAL0]
Annexe 6. Liste des actes de mastectomie permettant la facturation du GHS 3362,3363,3364 ou 3365. [AMAST]
Annexe 7 :
– liste 1. Actes marqueurs de suppléance vitale pour lesquels une occurrence suffit [AUSC1]
– liste 2. Actes marqueurs de suppléance vitale pour lesquels il faut au moins trois occurrences [AUSC3]
– liste 3. Actes de pose d’un dispositif d’assistance ventriculaire donnant lieu à rémunération du GHS 1518,1519,1520,1521,8934,8935,8936 ou 8937. [AASSV]
Annexe 8 :
– liste 1. Diagnostics et actes associés autorisant la facturation d’un supplément de surveillance continue [DAUSC]
– liste 2. Actes autorisant la facturation d’un supplément de surveillance continue [AUSCC]
– liste 3. Diagnostics et actes associés autorisant la facturation d’un supplément de surveillance continue pour les enfants de moins de 18 ans. [DAUSCP]
Annexe 9 :
– liste 1. Liste des GHS pouvant être facturés pour des prises en charge en hospitalisation de jour d’addictologie [GADDICTO]
– liste 2. Liste des activités justifiant la production d’un GHS d’addictologie lors d’une prise en charge de moins d’une journée. [AADDICTO]
Annexe 10. Liste des actes donnant lieu à rémunération sur la base d’un FFM. [AFFM]
Annexe 11. Liste des actes donnant lieu à rémunération sur la base des forfaits SE :
– liste 1. Liste des actes donnant lieu à rémunération sur la base d’un SE1 [ASE1]
– liste 2. Liste des actes donnant lieu à rémunération sur la base d’un SE2 [ASE2]
– liste 3. Liste des actes donnant lieu à rémunération sur la base d’un SE3 [ASE3]
– liste 4. Liste des actes donnant lieu à rémunération sur la base d’un SE4 [ASE4]
– liste 5. Liste des actes donnant lieu à rémunération sur la base d’un SE5 [ASE5]
– liste 6. Liste des actes donnant lieu à rémunération sur la base d’un SE6 [ASE6]
– liste 7. Liste des actes donnant lieu à rémunération sur la base d’un SE7 [ASE7]
Annexe 12. Liste des GHM et des diagnostics autorisant le supplément ante partum :
– liste 1. Liste des GHM autorisant le supplément ante partum [GANTEPART]
– liste 2. Liste des affections relatives à l’ante partum. [DANTEPART]
Annexe 13. Liste des actes donnant lieu à rémunération du GHS 7005. [AG7005]
Annexe 14. Liste des actes de détection isotopique donnant lieu à rémunération des GHS majorés “ Ganglion sentinelle ”. [AGGSI]
Annexe 15. Liste des actes d’anatomie pathologie du ganglion sentinelle donnant lieu à rémunération des GHS majorés “ Ganglion sentinelle ”. [AGGSP]
Annexe 16. Liste des actes donnant lieu à rémunération des GHS majorés “ rétine/ cataracte ”. [ACATAR]
Annexe 17. Liste des actes autorisation la facturation d’un supplément défibrillateur cardiaque. [ADEF]
Annexe 18. Liste des actes CCMU 2. [ACCMU2P]
Annexe 19. Liste des diagnostics :
– liste 1. Diagnostics pour lesquels la prise en charge au sein de la structure des urgences autorisée donne lieu à la facturation d’un supplément dénommé “ Supplément prise en charge pédiatrique ” (PE1). [DPE1]
– liste 2. Diagnostics pour lesquels la prise en charge au sein de la structure des urgences autorisée donne lieu à la facturation d’un supplément dénommé “ Supplément prise en charge pédiatrique ” (PE2). [DPE1]
Pour vous économiser de la saisie, voici un fichier CSV contenant l’équivalence :
Vous pouvez bien sûr changer les [noms] mais il faudra adapter tous les traitements que nous allons baser dessus.
Pour la suite de l’article il nous faudra quelques initialisations et librairies :
# On a déjà chargé la librairie de manipulation des fichiers excel
library(readxl)
# et il nous faudra aussi
library(dplyr)
library(readr)
library(tidyr)
library(stringr)
# on stocke la source dans une chaine de caractères pour la lisibilité future
f_source <- "https://www.atih.sante.fr/sites/default/files/public/content/4971/arrete_prestations_annexes_2025_vdef.xlsx"
# ici le nom de fichier de la table d'équivalence
f_equiv <- "./noms_annexes.csv"
# l'année à traiter
equiv_annee <- 2025
#Le chemin d'export des résultats
d_out <- sprintf("~/EXPORTS/REF/%4d/ARRETES/",equiv_annee)
Maintenant on est prêts, allons y !
Le chargement des données sources
Toujours grâce à la librairie readxl, nous allons cette fois-ci charger les tables brutes.
Ouvrir en lecture un fichier excel correspond à récupérer les données d’une (et une seule) des feuilles contenues dans celui-ci. Pour cela on utilise la fonction read_excel(). Comme souvent, il existe un bon nombre de paramètres, mais pour notre usage, nous allons nous contenter du strict minimum : path= qui représente le chemin vers le fichier et sheet= qui précise le nom de la feuille à récupérer. read_excel() n’est pas vectorisé, il n’est donc pas possible de récupérer en une fois plusieurs feuilles. D’ailleurs il se plaint :
> read_excel(fichier_existant, sheet = c("feuille1", "feuille2"))
Erreur : `sheet` must have length 1Qu’à cela ne tienne nous allons faire le job via un lapply() et en profiter pour changer ces noms bizarres :
# on charge la table d'équivalence
nom_equiv <- read_csv2(f_equiv) %>%
filter(ANNEE %in% equiv_annee & (is.na(DESACTIVE) | DESACTIVE==FALSE))
# on charge l'ensemble des annexes.
annexes <- nom_equiv %>% mutate(tbl = lapply(TABLE, FUN = \(x){
read_excel(path = f_temp, sheet = x)
})) %>%
{setNames(.$tbl, .$NOM)}
# à partir d'ici on possède dans "annexes" une liste de
# dataframes contenant l'ensemble des données brut d'import.La première ligne récupère le contenu de notre tableau d’équivalence de nom de feuilles et filtre pour ne traiter que celles qui seront pertinentes.
Le 2ème bloc se lit ainsi (en respectant les priorités d’exécution) :
- Prend le dataframe nom_equiv
- Ajoute une colonne tbl qui contiendra le résultat de la fonction se trouvant dans
FUN=appliquée au champ TABLE.- (Dans la fonction qui passe dans
x=une à une chaque valeur du champ TABLE ) - charge la feuille de nom x depuis le document f_temp.
- (Dans la fonction qui passe dans
- Dans un bloc dédié…
- Prend la colonne tbl nouvellement traitée (comme toute colonne d’un dataframe, c’est directement un objet de type liste), attribue un nom à chaque valeur en y mettant NOM en respectant l’ordre
- retourne cette liste sans modifier la source (implicite en fin de fonction)
- Charge le résultat dans l’object annexes (
<-)
Nous nous retrouvons donc avec, dans annexes, une liste nommée de dataframes. Il « suffit » d’appeler par exemple annexes$GHS pour récupérer les données correspondantes.
> annexes$GHS
# A tibble: 5,671 × 3
GHS GHM LIBELLÉ
<dbl> <chr> <chr>
1 22 01C031 Craniotomies pour traumatisme, âge supérieur à 17 ans, niveau 1
2 23 01C032 Craniotomies pour traumatisme, âge supérieur à 17 ans, niveau 2
3 24 01C033 Craniotomies pour traumatisme, âge supérieur à 17 ans, niveau 3
4 25 01C034 Craniotomies pour traumatisme, âge supérieur à 17 ans, niveau 4
5 26 01C041 Craniotomies en dehors de tout traumatisme, âge supérieur à 17 ans, niveau 1
6 27 01C042 Craniotomies en dehors de tout traumatisme, âge supérieur à 17 ans, niveau 2
7 28 01C043 Craniotomies en dehors de tout traumatisme, âge supérieur à 17 ans, niveau 3
8 29 01C044 Craniotomies en dehors de tout traumatisme, âge supérieur à 17 ans, niveau 4
9 30 01C051 Interventions sur le rachis et la moelle pour des affections neurologiques, niveau 1
10 31 01C052 Interventions sur le rachis et la moelle pour des affections neurologiques, niveau 2
# ℹ 5,661 more rows
# ℹ Use `print(n = ...)` to see more rowsVous noterez dans le code ci-dessus l’usage de setNames() pour fixer les noms mais aussi son usage entre { } et avec l’adressage de la source via « . » (cf l’article sur magrittr) qui permet en un temps de partir d’un dataframe (qui est l’argument devant le pipe %>%) pour arriver à la liste nommée qui nous intéresse.
Dans une perspective ETL, nous avons
- Extrait les données (la boucle de chargement),
- Transformé celles-ci (le
{setNames()}) - et avons sauvegardé (Load) dans une objet de type liste
Bravo, nous avons créé une source de données secondaire (certes peu transformées).
Cependant si vous regardez vos données récupérées, elles ne sont clairement pas propres. On peut noter plusieurs problèmes :
- Noms de colonnes non standardisés entre les tableaux
- code CIM10 non utilisables tels quels (avec des « . » en 4è position)
- Certaines colonnes inutiles et d’autres redondantes
- probablement un intérêt à n’avoir qu’une liste par GHS/GHM, Diagnostic, Acte ou Code avec des propriétés supplémentaires
Au boulot !
Les retraitements spécifiques
Je ne vais détailler que quelques tables, je vous laisserai généraliser à l’ensemble.
Les GHS
La table principale $GHS
La table « GHS » contient donc l’association GHS/GHM ainsi que le libellé de celui-ci. Il n’y a pas grand chose à changer. Je me contente de renommer la colonne LIBELLÉ en LIB. On pourrait se dire que c’est purement cosmétique mais cela à 2 intérêts : C’est plus court et aussi parlant, et surtout cela lève une classique source potentielle d’erreur de frappe en la présence d’un accent sur le « É » (je vous conseille fortement de proscrire les signes diacritiques dans les noms de variables !).
Dans la mesure où les données actuellement dans annexes seront mieux après, nous allons faire la sauvegarde « sur place », c’est à dire remplacer les données sources :
annexes$GHS <- annexes$GHS %>% rename(LIB = LIBELLÉ)En terme ETL : E : prend la table GHS / T : renomme LIBELLÉ en LIB / L : écrase la table GHS avec les nouvelles données
Les tables $GHSI et $GHSU
Ces deux tables sont des sous-sélections de la table GHS agrémentées de données complémentaires provenant de leur simple existence au sein de la table. Il serait donc plus pertinent de coupler ces données au sein d’une unique table en positionnant un champ précisant la donnée.
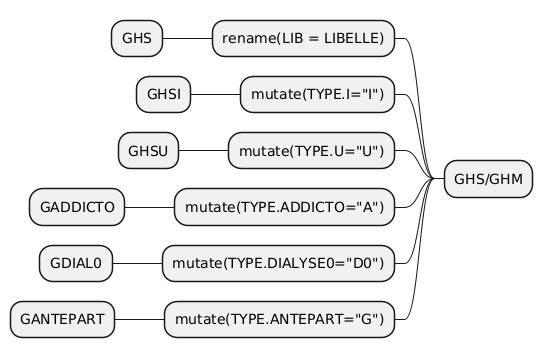
Au final, vu les données disponibles dans les annexes nous pouvons regrouper. Pour cela nous allons rajouter un ensemble de champs de typage.
Notre chaine ETL va donc être adaptée au niveau du T : ajout d’un champ spécifique avec le type et suppression du libellé. Puis une fois que chaque source sera faite nous les fusionnerons dans une table unique.
# La mise au propre
annexes$GHSI <- annexes$GHSI %>% mutate(TYPE.I = "I") %>% select(-LIBELLÉ)
annexes$GHSU <- annexes$GHSU %>% mutate(TYPE.U = "U") %>% select(-LIBELLÉ)
(...etc...)
# La fusion
GHSGHM <- annexes$GHS %>%
left_join(annexes$GHSI, by=c("GHS", "GHM")) %>%
left_join(annexes$GHSU, by=c("GHS", "GHM")) %>%
left_join(annexes$GADDICTO, by=c("GHS", "GHM")) %>%
left_join(annexes$GDIAL0, by=c("GHS","GHM")) %>%
left_join(annexes$GANTEPART, by=c("GHM")) %>%
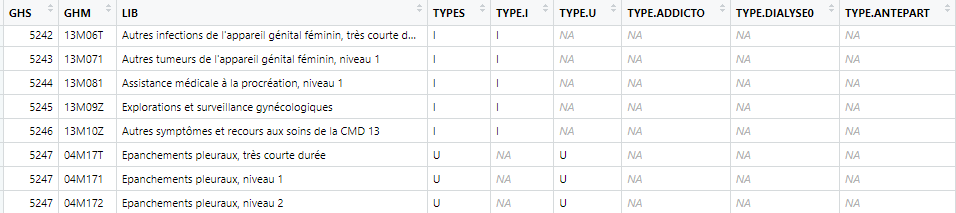
unite(col="TYPES", starts_with("TYPE."), na.rm=TRUE, remove=FALSE, sep="/")unite() (élément de tidyr) permet de regrouper le texte de plusieurs colonnes en une seule, le réglage de remove= à FALSE permet de garder aussi les colonnes initiales. starts_with() permet de sélectionner les colonnes commençant par « TYPE. »
Cette nouvelle table n’est pas per se dans les annexes, je ne l’insère donc pas dans annexes. Voici, par exemple son contenu pour quelques GHS :
On peut donc dire que pour produire la chaine ETL complète des GHSGHM, nous avons fait :
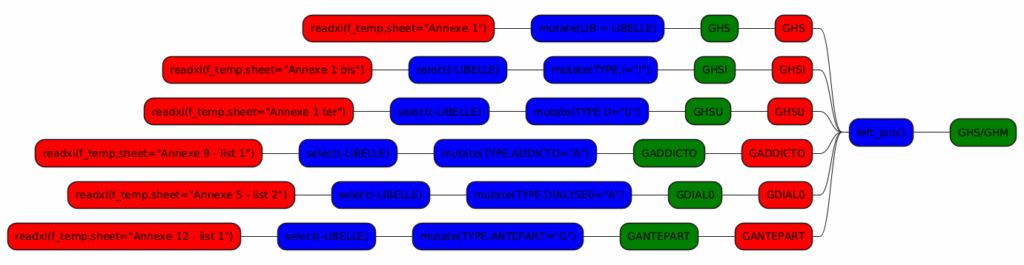
(libre à chacun d’extraire aussi les tables partielles (GHS, GHSI, GHSU, etc…) mais elles pourraient être recrées depuis GHSGHM par un simple filter(), par exemple:
> GHSGHM %>% filter(TYPE.I == "I") %>% select(1:3)
# A tibble: 307 × 3
GHS GHM LIB
<dbl> <chr> <chr>
1 348 01M04T Méningites virales, très courte durée
2 349 01M15T Accidents ischémiques transitoires et occlusions des artères précérébrales, âge supérieur à 79 ans, très courte durée
3 350 01M16T Accidents ischémiques transitoires et occlusions des artères précérébrales, âge inférieur à 80 ans, très courte durée
4 351 01M18T Lésions traumatiques intracrâniennes sévères, très courte durée
5 352 01M21T Douleurs chroniques irréductibles, très courte durée
6 353 01M27T Autres tumeurs du système nerveux, très courte durée
7 354 01M28T Hydrocéphalies, très courte durée
8 355 01M291 Anévrysmes cérébraux, niveau 1
9 356 01M30T Transferts et autres séjours courts pour accidents vasculaires intracérébraux non transitoires
10 357 01M31T Transferts et autres séjours courts pour autres accidents vasculaires cérébraux non transitoires
# ℹ 297 more rows
# ℹ Use `print(n = ...)` to see more rowsIl ne reste plus qu’à finaliser cette chaine en sauvegardant son résultat par exemple par un write_csv2(GHSGHM,"<le nom que vous désirez utiliser>"). A partir de là, il sera utilisable dans vos traitements par un read_csv2(...).
write_csv2(GHSGHM, file = paste(d_out,"GHSGHM.csv"), na = "")Rien ne vous empêche de faire d’autres retraitements, par exemple définir la clé de regroupement de GHM ou de GHS.
Les regroupements de GHM
Nous avons pour cela besoin de regrouper par racine de GHM :01C031 devient RGHM = 01C03 avec SEV =1.
Je vous mets directement la recette et j’ai commenté :
# L'aiguille permet de repérer la partie variant avec la sévérité dans le libellé
# afin de le neutraliser. Heureusement c'est assez standardisé...
aiguille_RGHM <- "(^.*)(, niveau.*|, très courte durée|, en ambulatoire|, avec autres complications|, avec complications majeures|, avec complications sévères|, sans complication significative)$"
RGHM <- annexes$GHS %>% # On aurait pû utiliser GHSGHM, cela revient au même
select(-GHS) %>% # Cette ligne n'est pas strictement nécessaire
# On fait l'extraction des données pertinentes
mutate(RGHM = substr(GHM, 1, 5), # les 5 1ers caractères du GHM
SEV = substr(GHM, 6, 6), # la sévérité sur 1 caractère
LIBR = gsub(aiguille_RGHM, "\\1", LIB)) %>% # le libellé
# reconstitué par restriction de l'aiguille
group_by(RGHM, LIBR, SEV) %>% # Simplification
summarise(n=n()) %>% # On en profite pour compter le nombre de GHS
# dans le RGHM et la sévérité
pivot_wider(names_from = "SEV",values_from = "n") # on en fait des colonnes
# Il est possible de faire des vérifications préliminaires
stopifnot("Il existe des RGHM à plus de 1 occurence" = all((RGHM %>% count(RGHM,LIBR))$n <=1))
write_csv2(RGHM,file = paste(d_out,"RGHM.csv"), na = "")Les regroupements de GHS
Si vous regardez la table annexes$GHS, vous remarquez que les GHM sont dédoublés mais que certains GHS le sont aussi. Là aussi, nous allons produire une liste plus propre :
RGHS <- annexes$GHS %>%
group_by(GHS) %>%
summarise(GHMs = paste(GHM, collapse = ","), # on crée un champ texte
# représentant une liste des GHM reliés au GHS
LIBR = paste(LIB, collapse = " / "), # on crée un champ texte
# reprenant les libellés de tous ces GHM
n = n(), # on garde le nombre en cas de besoin
TYPE.U = ifelse(all(GHS %in% annexes$GHSU$GHS),"U",NA),
TYPE.I = ifelse(all(GHS %in% annexes$GHSI$GHS),"I",NA)
# si tous les GHM contenus sont d'urgence ou intermédiaire on le
# signale dans une colonne à part.
) %>%
arrange(GHS) # et on trie par n° de GHS (facultatif bien sûr)
write_csv2(RGHS, file = paste(d_out,"RGHS.csv"), na = "")Vous en voulez encore ?
Les actes CCAM
Dans les annexes, il existe plusieurs tables se rapportant aux actes CCAM.
Nous n’allons pas importer la CCAM complète juste pour la retraiter mais simplement traiter les actes qui sont dans les annexes et qui sont les seuls pertinents. Il suffira lorsque le besoin s’en fera sentir de réutiliser le retraitement par un left_join() depuis une CCAM complète pour l’augmenter des données ici-présentes.
Comme pour les GHS nous allons produire une table composée de toutes les annexes en « A ».
D’abord retraiter les annexes
Il va nous falloir nous mettre dans un format homogène, pour cela on rajoute un champ « ACTE.XXXX » ou XXXX se réfère au contenu de la table, on supprime le champ libellé (qui sera récupéré en cas de besoin depuis une CCAM complète), on met le nom de la colonne du code CCAM à « CODE » et l’extension PMSI à « EXTPMSI » (pour éviter les espaces dans le nom de champ, pénibles à gérer).
Sur la première table d’annexes contenant des actes (les actes d’irradiation), cela nous donne ce pré-traitement :
annexes$ARAD1 <- annexes$ARAD1 %>% rename(EXTPMSI = `EXT. PMSI`) %>% mutate(ACTE.IRRAD = TRUE) %>% select(-3)
Pour changer, j’ai supprimé le libellé par position (-3) signifiant « supprime la 3ème colonne ».
La plupart des tables suivent ce type de nommage de colonnes SAUF que l’être humain étant ce qu’il est, certaines ne sont pas congruentes. Par exemple la table CCMU2P possède une ligne d’entête de plus, a un champ vide en première position et n’a pas de champ « EXT. PMSI ». Il faut remettre tout ça d’aplomb. Ce qui nous donne par exemple :
annexes$ACCMU2P <- annexes$ACCMU2P[2:nrow(annexes$ACCMU2P),] %>% mutate(ACTE.CCMU2P = TRUE) %>% select(-1,-3)
# On a pas créé le champ EXTPMSI qui n'a pas d'utilité propre
Je vous laisse faire les autres tables de ce type.
Fusionner les tables
Les SE
Pour la conformation des $ASE1..7, nous allons utiliser :
annexes$ASE1 <- annexes$ASE1 %>% rename(EXTPMSI=`EXT. PMSI`) %>% mutate(ACTE.SE1=TRUE,ACTE.SE="SE1") %>% select(-3)
C’est à dire que nous ne créons pas un ACTE.SE1, ACTE.SE2, etc… mais juste un ACTE.SE avec le « texte » du « SEn » correspondant.
Les SE sont particuliers car avoir plusieurs tables séparées n’est pas pertinent, d’une part ils s’excluent mutuellement et rechercher dans la table doit pouvoir répondre à la question « Quel SE pour mon acte CCAM ? » or si on garde des tables séparées, cela sera pénible et peu intuitif. Créons donc une table annexes@ASE qui va regrouper toutes les tables annexes@ASE1…7 (après que nous les ayons conformées comme ci-dessus) :
annexes$ASE <- bind_rows(annexes$ASE1,
annexes$ASE2,
annexes$ASE3,
annexes$ASE4,
annexes$ASE5,
annexes$ASE6,
annexes$ASE7) %>%
select(CODE, `EXTPMSI`, ACTE.SE)Fusionnons le tout
Maintenant que nous avons toutes nos tables d’actes dans un format compatible, nous pouvons fusionner le tout :
# On fusionne tout avec comme clé primaire le couple CODE/EXTPMSI
ACTES <- annexes$ARAD1 %>%
full_join(annexes$ADIAL1, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$AMAST, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$AUSC1, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$AUSC3, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$AASSV, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$AUSCC, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$AFFM, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$ASE, by=c("CODE","EXTPMSI")) %>%
full_join(annexes$AG7005, by=c("CODE","EXTPMSI"))%>%
full_join(annexes$AGGSI, by=c("CODE","EXTPMSI"))%>%
full_join(annexes$AGGSP, by=c("CODE","EXTPMSI"))%>%
full_join(annexes$ACATAR, by=c("CODE","EXTPMSI"))%>%
full_join(annexes$ADEF, by=c("CODE","EXTPMSI"))%>%
# sauf pour les actes CCMU2P qui n'ont pas le champ EXTPMSI
full_join(annexes$ACCMU2P, by=c("CODE"))Ce qui nous donne par exemple :
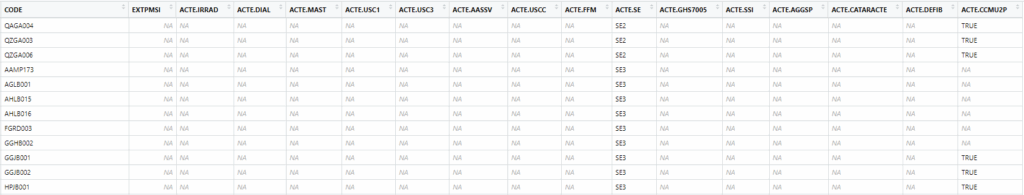
Et comme précédemment :
write_csv2(ACTES, file = paste(d_out,"ACTES.csv"), na = "")Les diagnostics
Les listes de diagnostic doivent aussi être conformées. En particulier, ces annexes sont prévues pour être lues par des humains, le format des codes CIM10 est donc l’écriture avec un point en 4ème position qu’il va falloir enlever pour tout traitement informatique. Nous allons aussi normaliser le nom de colonne « CODE » pour toutes les colonnes contenant ce type de données.
annexes$DANTEPART <- annexes$DANTEPART %>%
mutate(CODE = str_remove(`CODES`, "\\."), .keep="none")
# %>% select(CODE)
Notez l’utilisation de str_remove() pour enlever tous les « . » présents dans la chaine source (il n’y en a qu’un normalement) et aussi de l’option .keep= "none" dans le mutate() qui va créer CODE et dans le même temps supprimer tous les champs (en ne gardant (keep) que les champs créés). En théorie le %>% select(CODE) n’est pas nécessaire grâce à ce réglage.
Les valeurs possible de .keep= sont :
Les forfaits
Ces tables sont peut intéressantes, elles listent juste les forfaits existants. On va passer rapidement.
Mise au format :
annexes$FDIAL <- annexes$FDIAL %>% rename(LIB = FORFAIT) %>% mutate(TFORFAIT = "DIALYSE")
annexes$POE <- annexes$POE %>% rename(LIB = FORFAIT) %>% mutate(TFORFAIT = "POE")
annexes$POP <- annexes$POP %>% rename(LIB = FORFAIT) %>% mutate(TFORFAIT = "POP")Et concaténation :
FORFAITS <- bind_rows(annexes$FDIAL,
annexes$POE,
annexes$POP
)Sauvegarde :
write_csv2(ACTES, file = paste(d_out,"ACTES.csv"), na = "")
Conclusion
Nous avons désormais une procédure indépendante pour créer des tables pertinentes représentant l’arrêté « Prestations ». La prochaine fois nous allons voir l’arrêté « Tarifaire ».
D’ici-là essayez de reprendre vos traitements avec cette approche ETL qui permet de planifier proprement vos pré-traitements et de les réutiliser d’un projet à l’autre.

