S’il y a bien une star parmi les fichiers PMSI MCO c’est le RSS. Celui-ci liste les RUM de tous les séjours exportés.
Dans cet article nous allons aborder non pas sa découpe propre (le principe est le même que le fichier des UM) dans l’article Le découpage de fichiers “à plat” mais plutôt aborder le retraitement, le regroupement et essayer de tirer quelques conclusions de ce fichier.
Pour que vous puissiez « jouer » avec un jeu de données qui vous est propre et pertinent, je vais me baser sur le format MCO 2021 qui sera à alimenter avec des données 2021 (les années récentes devraient en fait toutes passer vu que nous ne cherchons pas à être exhaustif sur l’import). Vous pourrez ainsi injecter le RSS de votre établissement.
Préparation
Pour lire le RSS, il va nous falloir readr et pour les manipulations, dplyr comme pour le fichier des UM. Nous allons aussi aborder une fonction qui se trouve dans tidyr.
library(dplyr) # dplyr implique magrittr mais on pourrait le spécifier en plus sans conséquence.
library(readr)
library(tidyr)
Il va aussi nous falloir un ficher de RSS groupé (format 120) que j’ai appelé chez mois « DEF.RSS ».
Nous allons lire les champs du RSS groupé qui nous intéressent et leur donner un nom. J’ai décidé de ne récupérer que (entre crochets les positions dans la ligne du RSS) :
- GHMC [3 – 8]: « complet » qui contient le GHM du RSS groupé, j’en profite pour extraire
- SEV [8 – 8]: le niveau de sévérité du GHM
- RGHM [3 – 7]: le GHM sans le niveau de sévérité
- CMD [3 – 4]: la catégorie majeur de diagnostic
- NRSS [28 – 47]: numéro de RSS
- NRUM [68 – 77]: numéro de RUM
- NDA [48 – 67]: numéro d’entrée propre à l’établissement
- SEXE [86 – 86]
- DN [78 – 85] : date de naissance
- DENT [93 – 100]: date d’entrée
- DSORT [103 – 110]: date de sortie
- MENT [101 – 101]: mode d’entrée
- CENT [102 – 102]: provenance (je mets CENT car je trouve cela plus parlant que « provenance »)
- MSORT [111 – 111]: mode de sortie
- CSORT [112 – 112]: le complément au mode de sortie (même remarque que ci-dessus)
- UM [87 – 90]: code de l’unité médicale
Ce qui nous donne le code suivant :
rss <- read_fwf(file = "DEF.RSS",
col_positions = fwf_cols(GHMC = c(3, 8),
SEV = c(8, 8),
RGHM = c(3, 7),
CMD = c(3, 4),
NRSS = c(28, 47),
NRUM = c(68, 77),
NDA = c(48, 67),
SEXE = c(86, 86),
DN = c(78, 85),
DENT = c(93, 100),
DSORT= c(103, 110),
MENT = c(101, 101),
CENT = c(102, 102),
MSORT= c(111, 111),
CSORT= c(112, 112),
UM = c(87,90)
),
col_types = paste(rep("c", times = 15), collapse = "")
)
Ne voulant pas m’embêter, j’ai tout récupéré en colonne de chaines de caractères grâce à la définition de col_types à 16 fois le caractère ‘ »c ».
paste(rep("c", times = 16), collapse = "")
est à comprendre comme crée un vecteur où répète 16 fois le caractère « c », puis comprime se vecteur dans une chaine de caractère avec <rien> (« ») entre chaque. Ce qui donne bien « ccccccccccccccc ». L’utilisation de paste pour coller des chaines de caractères ensemble n’est pas super intuitif mais on s’y fait vite.
L’exécution de ce code va charger votre fichier de RSS dans le jeu de données « rss » que vous pouvez voir dans la fenêtre des données en haut à droite.
Et en cliquant sur la ligne vous pouvez consulter son contenu dans la tuile en haut à gauche :
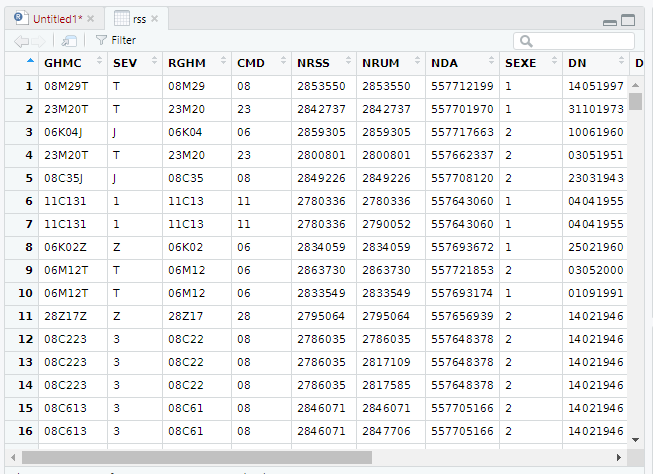
Comme vous pouvez le voir, mon découpage n’a pas à être dans l’ordre ou à ne pas se superposer. En une seule passe, j’extrais par exemple le GHMC, RGHM, SEV et CMD qui sont sur les mêmes indexes sur la ligne.
Reste le problème des dates, mais nous avons vu dans l’article Le découpage de fichiers “à plat” comment faire. Nous complétons donc par les mutate qui vont bien :
rss <- read_fwf("~/EXPORTS/PESSAC/2021/DEF.RSS",
col_positions = fwf_cols(GHMC = c(3, 8),
SEV = c(8, 8),
RGHM = c(3, 7),
CMD = c(3, 4),
NRSS = c(28, 47),
NRUM = c(68, 77),
NDA = c(48, 67),
SEXE = c(86, 86),
DN = c(78, 85),
DENT = c(93, 100),
DSORT= c(103, 110),
MENT = c(101, 101),
CENT = c(102, 102),
MSORT= c(111, 111),
CSORT= c(112, 112),
UM = c(87,90)
),
col_types = paste(rep("c", times = 16), collapse = "")
) %>%
mutate( DN = as.Date(DN, format = "%d%m%Y"),
DENT = as.Date(DENT, format = "%d%m%Y"),
DSORT = as.Date(DSORT, format = "%d%m%Y")
)
Puis pour compléter, nous alors importer de même le fichier des UM vu dans l’article Le découpage de fichiers “à plat”.
En résumé le code complet permettant de charger les données est :
library(dplyr)
library(readr)
rss <- read_fwf("DEF.RSS",
col_positions = fwf_cols(GHMC = c(3, 8),
SEV = c(8, 8),
RGHM = c(3, 7),
CMD = c(3, 4),
NRSS = c(28, 47),
NRUM = c(68, 77),
NDA = c(48, 67),
SEXE = c(86, 86),
DN = c(78, 85),
DENT = c(93, 100),
DSORT= c(103, 110),
MENT = c(101, 101),
CENT = c(102, 102),
MSORT= c(111, 111),
CSORT= c(112, 112),
UM = c(87,90)
),
col_types = paste(rep("c", times = 16), collapse = "")
) %>%
mutate( DN = as.Date(DN, format = "%d%m%Y"),
DENT = as.Date(DENT, format = "%d%m%Y"),
DSORT = as.Date(DSORT, format = "%d%m%Y")
)
ums <- read_fwf( file = "DEF.UM",
col_positions = fwf_cols(UM = c(1, 4),
FINESS = c(5, 13),
AUTH = c(14, 16),
DDEB = c(17, 24),
NLITS = c(25, 27),
MODE = c(28,28)
),
col_types="ccccnc"
) %>% mutate( DDEB = as.Date(DDEB, format = "%d%m%Y"))
Ce RSS va nous servir à expérimenter dplyr. Martyrisons-le un peu.
Ajoutons des colonnes calculées.
Comme vous vous en doutez probablement cela se fait avec mutate.
Si nous voulons calculer la durée de séjour dans l’UM (DRUM : durée du RUM). Il s’agit de la différence entre DSORT et DENT. On écrit donc :
> rss %>% mutate(DRUM = DSORT - DENT) %>% select(NDA,NRUM,DRUM)
# A tibble: 24,671 x 3
NDA NRUM DRUM
<chr> <chr> <drtn>
1 557712199 2853550 1 days
2 557701970 2842737 0 days
3 557717663 2859305 0 days
4 557662337 2800801 0 days
5 557708120 2849226 0 days
6 557643060 2780336 1 days
7 557643060 2790052 1 days
8 557693672 2834059 0 days
9 557721853 2863730 1 days
10 557693174 2833549 0 days
# ... with 24,661 more rows
(le select est là uniquement pour la présentation)
et voilà, toutes les lignes du RSS disposent maintenant de leur durée de séjour ! Notez bien cependant que le changement n’est pas permanent car le jeu de données n’a pas été réattribué à rss. Si nous voulons garder la donnée pour les traitements futurs, il faut rajouter un rss <- en début de ligne ce qui donne :
rss <- rss %>% mutate(DRUM = DSORT - DENT)
et si nous comptons systématiquement calculer DRUM pour tous nos RSS que nous ouvrons, on peut alors rajouter la colonne dès l’ouverture du fichier :
rss <- read_fwf("DEF.RSS",
col_positions = fwf_cols(GHMC = c(3, 8),
SEV = c(8, 8),
RGHM = c(3, 7),
CMD = c(3, 4),
NRSS = c(28, 47),
NRUM = c(68, 77),
NDA = c(48, 67),
SEXE = c(86, 86),
DN = c(78, 85),
DENT = c(93, 100),
DSORT= c(103, 110),
MENT = c(101, 101),
CENT = c(102, 102),
MSORT= c(111, 111),
CSORT= c(112, 112);
UM = c(87,90)
),
col_types = paste(rep("c", times = 16), collapse = "")
) %>%
mutate( DN = as.Date(DN, format = "%d%m%Y"),
DENT = as.Date(DENT, format = "%d%m%Y"),
DSORT = as.Date(DSORT, format = "%d%m%Y")
DRUM = DSORT - DENT)
Par contre, assez logiquement l’ordre des mutations est important, il n’est pas possible de calculer DRUM tant que DSORT et DENT ne sont pas des dates d’où sa position dans le mutate.
Croisons des données
dplyr donne des fonctions simples proche de SQL à nouveau pour croiser 2 jeux de données. Il s’agit de la famille des « *_join ».
Le prototype d’une fonction « *_join » est toujours le même :
jeu1 %>% *_join(jeu2, by = c("col1" = "col2"))
Où jeu1 et jeu2 sont 2 jeux de données et donc le contenu de col1 chez jeu1 et col2 chez jeu2 sont compatibles. Un paramètre complémentaire utile est suffix = c("chaine1","chaine2") qui permet de spécifier la façon dont va se comporter le programme en cas d’homonymie de colonne entre jeu1 et jeu2 si celles-ci ne font pas partie de la jointure. En l’absence de suffix défini la valeur par défaut est « .x » pour le jeu1 et « .y » pour le jeu2. Dans le paramètre by=, seul le « = » est autorisé (pas d’inégalité).
Il peut y avoir plusieurs colonnes de jointure que l’on sépare par des virgules (vous remarquez que c’est une concaténation donc un vecteur d’égalités) :
jeu1 %>% *_join(jeu2,by = c("col1.1" = "col2.1", "col1.2" = "col2.2"))
pour le by =, il existe une forme réduite si les colonnes ont le même nom dans les 2 jeux :
jeu1 %>% *_join(jeu2, by = "col1")
Il y a 2 principaux types de jointures :
- Les jointures mutatrices (qui modifient les colonnes du jeu de données en ajoutant à jeu1, les colonnes de jeu2 en dehors de celles de jointure)
- inner_join : qui produit un nouveau jeu de données contenant uniquement les observations ou col1=col2 est réalisable
- left_join/right_join : qui produit un jeu de données contenant toutes les valeurs gauche (jeu1) ou droite (jeu2) et leur pendant dans l’autre jeu. Les données absentes étant mises à
NA - full_join : qui assez logiquement produit un nouveau jeu avec toutes les données de jeu1 et jeu2 et des
NApour toutes les données manquantes de jeu1 ET jeu2.
- Les jointures de filtre (qui filtrent le jeu de données initial sans en modifier les colonnes)
- semi_join : qui limite jeu1 aux observations ayant des données dans jeu2
- anti_join : qui limite jeu1 aux observations n’ayant pas des données dans jeu2
Exemples :
Nous voulons vérifier que chaque RUM possède une UM dans le fichier des UM.
rss %>% anti_join(ums, by = c("UM" = "UM"))
Mon fichier d’UM étant volontairement un peu vieux, ma requête retourne des erreurs :
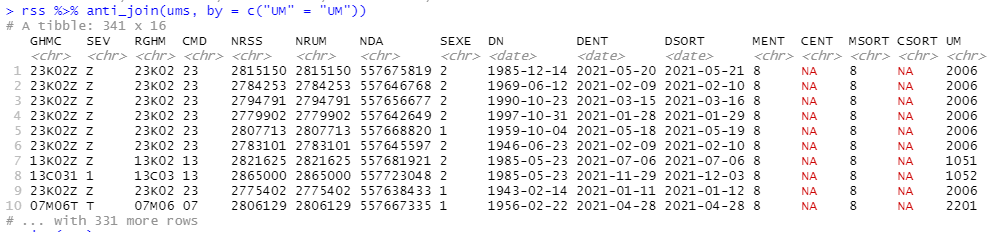
Je sais donc que j’ai 341 RSS faux.
Sauf que ce n’est pas le RSS qui est faux, mais les UM. Il nous faut donc isoler les UM en cause. On pourrait donc faire simplement:
rss %>% anti_join(ums, by = c("UM" = "UM")) %>% select(UM) %>% unique
qui se lit : prend rss, garde les lignes qui n’ont pas d’équivalent dans ums en joignant par UM, limite-toi à la colonne UM et ne garde qu’une observation par valeur d’UM.
Cela nous donne alors :
> rss %>% anti_join(ums, by = c("UM" = "UM")) %>% select(UM) %>% unique
# A tibble: 8 x 1
UM
<chr>
1 2006
2 1051
3 1052
4 2201
5 1151
6 1152
7 1402
8 1401
Voilà, nous savons quelles UM sont mal renseignées dans le fichier des UM. Il suffit de corriger la structure de l’établissement et de recharger les données corrigées dans ums pour refaire le contrôle.
Cependant, étant un peu en retard, nous préfèrerions travailler en priorité l’UM qui génère le plus d’erreur.
Il faut pour cela utiliser les fonctions de regroupement.
Les fonctions de regroupement
A nouveau, ces fonctions ont un air de SQL, elles correspondent à la logique derrière « GROUP BY » et s’appellent group_by() et summarise() (A noter que R est sympa, on peut l’écrire à l’anglaise ou à l’américaine summarize()).
Je vous introduis aussi arrange() qui permet de classer (comme « ORDER BY » en SQL)
La logique est « A tout group_by(), je joins un summarise()« .
Ainsi notre interrogation sur les UMs manquantes peut devenir
> rss %>% anti_join(ums, by = c("UM" = "UM")) %>%
group_by(UM) %>%
summarise(Nombre = n()) %>%
arrange(-Nombre)
# A tibble: 8 x 2
UM Nombre
<chr> <int>
1 2006 119
2 1051 65
3 1151 56
4 2201 44
5 1152 34
6 1402 13
7 1052 9
8 1401 1
Qu’on lit « (…) puis groupe par UM, au sein du groupe calcule le nombre d’occurrences et enfin affiche le résultat en triant par ordre décroissant de la valeur calculée « Nombre » « .
Les tableaux croisés
Si Excel est bien utilisé à quelque chose, c’est à faire des tableaux croisés car c’est une façon simple de visualiser des populations de cas selon 2 variables ou plus.
R fait de même grâce à des fonctions de la librairie tidyr, elle aussi membre du tidyverse (à son origine même). tidyr est une librairie elle-aussi très étoffée mais nous n’allons parler pour le moment que de la famille des fonctions pivot.
Supposons que nous voulions croiser le mode d’entrée et de sortie d’une UM (au hasard « 2002 »)
> rss %>% filter(UM == "2002") %>% group_by(MENT, MSORT) %>% summarise(Nombre = n())
# A tibble: 12 x 3
# Groups: MENT [3]
MENT MSORT N
<chr> <chr> <int>
1 6 6 67
2 6 7 60
3 6 8 354
4 6 9 55
5 7 6 13
6 7 7 17
7 7 8 42
8 7 9 3
9 8 6 36
10 8 7 88
11 8 8 496
12 8 9 41
Pas très lisible…
Il suffit ensuite de faire pivoter la colonne MSORT par exemple, grâce à la fonction pivot_wider :
> rss %>% filter(UM == "2002") %>% group_by(MENT, MSORT) %>% summarise(Nombre = n()) %>% pivot_wider(names_from = MSORT, values_from = Nombre)
`summarise()` has grouped output by 'MENT'. You can override using the `.groups` argument.
# A tibble: 3 x 5
# Groups: MENT [3]
MENT `6` `7` `8` `9`
<chr> <int> <int> <int> <int>
1 6 67 60 354 55
2 7 13 17 42 3
3 8 36 88 496 41
Et nous avons notre tableau croisé. Il est possible de croiser par plusieurs variables, ainsi pour affiner un peu plus par exemple en prenant aussi les champs provenance (CENT) et destination (CSORT)
> rss %>% filter(UM == "2002") %>%
group_by(MENT, CENT, MSORT, CSORT) %>%
summarise(Nombre = n()) %>%
pivot_wider(names_from = c(MSORT,CSORT), values_from = Nombre)
`summarise()` has grouped output by 'MENT', 'CENT', 'MSORT'. You can override using the `.groups` argument.
# A tibble: 6 x 9
# Groups: MENT, CENT [6]
MENT CENT `6_1` `6_2` `7_1` `7_2` `8_NA` `9_NA` `8_7`
<chr> <chr> <int> <int> <int> <int> <int> <int> <int>
1 6 1 23 39 26 33 352 50 NA
2 6 2 2 3 1 NA 2 5 NA
3 7 1 8 3 8 6 41 2 NA
4 7 2 1 1 NA 3 1 1 NA
5 8 5 11 9 63 9 274 27 NA
6 8 NA 8 8 9 7 221 14 1
Des NA ont été rajoutés, il est possible de les forcer à 0 en rajoutant l’option values_fill = 0 à pivot_wider.
On se retrouve alors avec un tableau croisé assez présentable :
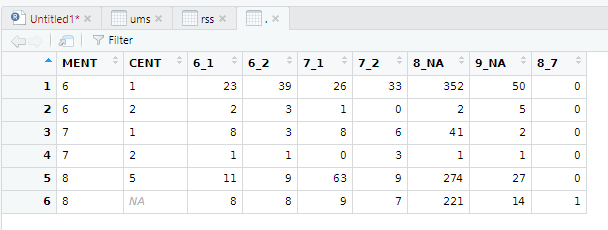
> rss %>% filter(UM == "2002") %>% group_by(MENT, CENT, MSORT, CSORT) %>% summarise(Nombre = n()) %>% pivot_wider(names_from = c(MSORT,CSORT), values_from = Nombre, values_fill = 0)
`summarise()` has grouped output by 'MENT', 'CENT', 'MSORT'. You can override using the `.groups` argument.
# A tibble: 6 x 9
# Groups: MENT, CENT [6]
MENT CENT `6_1` `6_2` `7_1` `7_2` `8_NA` `9_NA` `8_7`
<chr> <chr> <int> <int> <int> <int> <int> <int> <int>
1 6 1 23 39 26 33 352 50 0
2 6 2 2 3 1 0 2 5 0
3 7 1 8 3 8 6 41 2 0
4 7 2 1 1 0 3 1 1 0
5 8 5 11 9 63 9 274 27 0
6 8 NA 8 8 9 7 221 14 1
Ou en vue table :
Bien entendu, cette fonction ne faisant que transformer un jeu de données en un autre jeu de données pivoté, il est tout à fait possible de continuer à appliquer des filtres et transformation comme précédemment.
PS : pour voir un résultat de façon tabulaire graphique comme ci-dessus, il suffit de le « piper » dans la fonction View (avec un V majuscule) : rss %>% filter(UM == "2002") %>% group_by(MENT, CENT, MSORT, CSORT) %>% summarise(Nombre = n()) %>% pivot_wider(names_from = c(MSORT,CSORT), values_from = Nombre, values_fill = 0) %>% View
Faisons encore un peu de contrôle qualité pour illustrer
Nous allons continuer à jouer avec ces 2 jeux de données et faire un peu plus de contrôle qualité. C’est sur l’expressivité des fonctions de dplyr qu’on voit toute la supériorité par rapport à l’utilisation d’un tableur : R permets de faire très facilement du contrôle qualité sans devoir triturer à la main le jeu de données. On programme les mutations nécessaires puis on filtre, on fait un tableau de rapport et le contrôle est fini.
Cohérence des dates d’entrée et sortie
Nous voulons vérifier que tous les RUM possèdent une date de sortie supérieure ou égale à la date d’entrée. Le morceau de code ci-dessous nous listerait les RUMs correspondants aux erreurs.
> rss %>% filter(DSORT < DENT)
Et s’il y en avait beaucoup, on pourrait rajouter %>% nrow pour avoir leur nombre (nrow compte le nombre d’observations dans un jeu de données)
> rss %>% filter(DSORT < DENT) %>% nrow # doit valoir 0 !
[1] 0
Youpi, mon RSS est bon sur ce point, pfiuu.
Les UHCD bien tous rentrés par les urgences ?
Chez moi l’UHCD a le n° d’UM 3505. Le mode d’entrée « domicile par les urgences » est 8-5. Je pourrais donc faire
rss %>% filter( UM == "3505" & !(MENT == "8" & CENT == "5"))
Sauf que ce n’est pas très polyvalent car le code UM peut être différent, ou il peut y avoir plusieurs service d’UHCD. La solution ? Utiliser le fichier des UM grâce à une jointure :
rss %>% inner_join(ums %>% filter(AUTH = "07"),
by = "UM") %>%
filter(!(MENT == "8" & CENT == "5"))
Ainsi nous nous retrouvons avec uniquement les séjours dans une UM avec autorisation d’UHCD sans devoir spécifier le numéro de chaque UM.
Exercices
Je vous laisse expérimenter et vous propose quelques exercices :
- Isoler les séjours multiRUM et compter leur nombre. Classer de façon décroissante en fonction du nombre de RUM.
rss %>% group_by(NDA) %>%
summarise(MultiRUM = n()) %>%
group_by(MultiRUM) %>%
summarise(Nombre=n()) %>%
arrange(-Nombre)
# A tibble: 8 x 2
MultiRUM Nombre
<int> <int>
1 1 21168
2 2 1251
3 3 253
4 4 39
5 5 13
6 6 1
7 7 1
8 8 1- Générer une liste de séjours et la stocker dans le jeu de données sejours
sejours <- rss %>%
group_by(NDA) %>%
arrange(DENT,DSORT) %>%
summarise(DENT = first(DENT),
DSORT = last(DSORT),
MultiRUM = n(),
MENT = first(MENT),
CENT = first(CENT),
MSORT = last(MSORT),
CSORT = last(CSORT),
CMD = first(CMD),
SEV = first(SEV),
GHMC = first(GHMC),
RGHM = first(RGHM)
)- Isoler les sejours monorum de plus de 30 jours et faire une synthèse par CMD et sévérité
> sejours %>% filter(MultiRUM == 1 &
(DSORT - DENT >= 30)) %>%
group_by(CMD,SEV) %>%
summarise(Nombre = n()) %>%
arrange(CMD,SEV)
`summarise()` has grouped output by 'CMD'. You can override using the `.groups` argument.
# A tibble: 13 x 3
# Groups: CMD [9]
CMD SEV Nombre
<chr> <chr> <int>
1 01 2 1
2 01 3 1
3 04 3 1
4 04 4 1
5 05 4 2
6 07 4 3
7 08 3 1
8 08 4 2
9 11 2 1
10 11 4 1
11 17 3 1
12 19 3 1
13 23 Z 9- En faire un tableau croisé
> sejours %>% filter(MultiRUM == 1 &
(DSORT - DENT >= 30)) %>%
group_by(CMD,SEV) %>%
summarise(Nombre = n()) %>%
arrange(CMD,SEV) %>%
pivot_wider(names_from = SEV,
values_from = Nombre,
values_fill = 0)
`summarise()` has grouped output by 'CMD'. You can override using the `.groups` argument.
# A tibble: 9 x 5
# Groups: CMD [9]
CMD `2` `3` `4` Z
<chr> <int> <int> <int> <int>
1 01 1 1 0 0
2 04 0 1 1 0
3 05 0 0 2 0
4 07 0 0 3 0
5 08 0 1 2 0
6 11 1 0 1 0
7 17 0 1 0 0
8 19 0 1 0 0
9 23 0 0 0 9Conclusion
Ca y est nous avons atteint le Saint Graal, dompter un minimum le RSS. dans le prochain épisode nous allons écrire nos propres fonctions avant de continuer à décortiquer des champs plus ardus à récupérer.

