Le PMSI utilise principalement des fichiers que je qualifie de « à plat ». C’est à dire qu’il s’agit de fichiers textes dont les « observations » (au sens R) sont délimitées par des retours à la ligne mais chaque observation est dans un format fixe normé (dans les fameux fichiers excel de l’ATIH, par exemple format_mco2022.xlsx).
Pour notre exemple du jour nous allons prendre le cas du fichier IUM tout simplement car c’est celui qui contient le moins de colonnes.
Un fichier IUM typique contient 1 ligne par service et ressemble à ca :
2055990780529 01012020 C
110199078052911 01012020005M
350599078052907 01012020 C
120199078052911 01012020 M
325299078052903 01012020 C
225199078052911 01012020 P
205199078052911 01012020 P
1002990780529 01012020 CPour un oeil aguerri, on peut retrouver la structure :
110199078052911 01012020005M
1101 = code de l'UM
990780529 = FINESS géographique
11 = code d'autorisation
01012020 = date d'effet de l'autorisation au format préféré de l'ATIH "jjmmaaaa"
5 = Nombre de lits dans l'UM
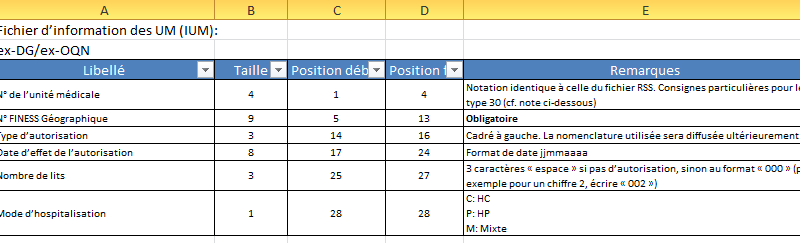
M = mode d'hospitalisation (M = mixte, P = partielle, C = complète)La lecture du fichier excel de l’ATIH nous donne les définitions officielles ainsi que les tailles et positions de découpe :
Passons aux choses sérieures
Commençons par charger les libraires incontournables
library(dplyr)
library(magrittr)
et ajoutons-y la librairie readr, qui comme les précédentes fait partie du tidyverse
library(readr)
Readr donne accès à plusieurs fonctions de lecture de fichiers selon des formats variables.
Celle qui nous intéresse est read_fwf qui permet de lire des formats à champs de longueur prédéfinie (Fixed Width Field)
Si on regarde l’aide de la fonction, on trouve :
read_fwf(
file,
col_positions,
col_types = NULL,
locale = default_locale(),
na = c("", "NA"),
comment = "",
trim_ws = TRUE,
skip = 0,
n_max = Inf,
guess_max = min(n_max, 1000),
progress = show_progress(),
skip_empty_rows = TRUE
)Nous devons donc fournir au minimum, un nom de fichier (file) et un ensemble de positions (col_positions) pour le découpage. Les autres paramètres sont optionnels.
En se basant sur format_mco2022 et le mode d’emploi de read_fwf, on en déduit un contenu possible de col_positions :
fwf_cols(UM = c(1, 4), FINESS = c(5, 13), AUTH = c(14, 16), DDEB = c(17, 24), NLITS = c(25, 27), MODE = c(28,28))Ce qui nous donne
read_fwf("fich.UM",fwf_cols(UM = c(1, 4), FINESS = c(5, 13), AUTH = c(14, 16), DDEB = c(17, 24), NLITS = c(25, 27), MODE = c(28,28)))
ou pour gagner en lisibilité :
ums <- read_fwf( file = "fich.UM",
col_positions = fwf_cols(UM = c(1, 4),
FINESS = c(5, 13),
AUTH = c(14, 16),
DDEB = c(17, 24),
NLITS = c(25, 27),
MODE = c(28,28)
)
)On voit bien apparaître les données de positionnement telles qu’elles sont dans format_mco.
Pas encore tout à fait ça
Si nous lançons la commande ci-dessus, nous nous retrouvons avec un jeu de données
# A tibble: 32 x 6
UM FINESS AUTH DDEB NLITS MODE
<dbl> <dbl> <chr> <chr> <chr> <chr>
1 2055 990780529 NA 01012020 NA C
2 1101 990780529 11 01012020 5 M
3 3505 990780529 07 01012020 NA C
4 1201 990780529 11 01012020 NA M
5 3252 990780529 03 01012020 NA C
6 2251 990780529 11 01012020 NA P
7 2051 990780529 11 01012020 NA P
8 1002 990780529 NA 01012020 NA C
9 1001 990780529 11 01012020 NA M
10 2451 990780529 11 01012020 NA P
# ... with 22 more rows
Un problème se pose cependant, c’est le type de données de chaque colonne. Readr a essayé de deviner ! Il a décidé que UM et FINESS étaient numériques et le reste des chaines de caractères. Pour cela il étudie environ 1000 lignes et cherche le type qui correspond à toutes, le type character étant celui acceptant tout.
NA représente des données non disponibles, dans le cas présent un champ « vide » (ou plutôt composé uniquement d’espaces).
Or cela ne nous va pas. NLITS est obligatoirement numérique, DDEB est une date et n’ayant pas de calculs à faire dessus, il est préférable que UM et FINESS soient des chaines de caractères.
Pour régler le problème de NLITS, on pourrait demander à read_fwf de faire lui même la correction de type en précisant le paramètre col_types . Il suffit d’y indiquer une chaine de caractère détaillant les types de chaque champ ainsi si nous voulons chaine-chaine-chaine-chaine-numérique-chaine, on assigne « cccnc » à col_types (je le fais dans la fenêtre exécution):
> read_fwf( file = "DEF.UM",
+ col_positions = fwf_cols(UM = c(1, 4),
+ FINESS = c(5, 13),
+ AUTH = c(14, 16),
+ DDEB = c(17, 24),
+ NLITS = c(25, 27),
+ MODE = c(28,28)
+ ),
+ col_types="ccccnc"
+ )
# A tibble: 32 x 6
UM FINESS AUTH DDEB NLITS MODE
<chr> <chr> <chr> <chr> <dbl> <chr>
1 2055 990780529 NA 01012020 NA C
2 1101 990780529 11 01012020 5 M
3 3505 990780529 07 01012020 NA C
4 1201 990780529 11 01012020 NA M
5 3252 990780529 03 01012020 NA C
6 2251 990780529 11 01012020 NA P
7 2051 990780529 11 01012020 NA P
8 1002 990780529 NA 01012020 NA C
9 1001 990780529 11 01012020 NA M
10 2451 990780529 11 01012020 NA P
# ... with 22 more rows
Reste le problème de la date qui n’est pas au format standard (R utilise le format aaaa-mm-dd par défaut). Malheureusement, même si il existe la lecture directe au format date grâce au caractère « D » dans le col_types, cela ne se passe pas bien :
Warning: 32 parsing failures.
row col expected actual file
1 DDEB date like 01012020 'DEF.UM'
2 DDEB date like 01012020 'DEF.UM'
3 DDEB date like 01012020 'DEF.UM'
4 DDEB date like 01012020 'DEF.UM'
5 DDEB date like 01012020 'DEF.UM'
... .... .......... ........ ........
See problems(...) for more details.
Nous allons donc faire appel à une fonction de dplyr pour corriger ce type : mutate et à la fonction de formatage as.Date.
mutate permet de définir de nouvelles colonnes ou de redéfinir des colonnes existantes à partir de valeurs du jeu de données ou des variables du programme.
as.Date modifie le type de données et dans le cas présent va nous servir à transformer une chaine de caractère en date au format R. Nous savons que R ne sait pas comment lire cette date donc nous allons devoir lui expliquer en précisant le format via le paramètre du même nom
> as.Date("02012020", format = "%d%m%Y")
[1] "2020-01-02"
C’est bon le format est le bon. Il suffit de reformater l’ensemble de la colonne DDEB grâce à mutate
ums %>% mutate(DDEB = as.Date(DDEB, format = "%d%m%Y")
# A tibble: 32 x 6
UM FINESS AUTH DDEB NLITS MODE
<chr> <chr> <chr> <date> <dbl> <chr>
1 2055 990780529 NA 2020-01-01 NA C
2 1101 990780529 11 2020-01-01 5 M
3 3505 990780529 07 2020-01-01 NA C
4 1201 990780529 11 2020-01-01 NA M
5 3252 990780529 03 2020-01-01 NA C
6 2251 990780529 11 2020-01-01 NA P
7 2051 990780529 11 2020-01-01 NA P
8 1002 990780529 NA 2020-01-01 NA C
9 1001 990780529 11 2020-01-01 NA M
10 2451 990780529 11 2020-01-01 NA P
# ... with 22 more rows
et il suffit de le réattribuer à lui-même pour que notre table soit correcte. Ni vu ni connu.
ums <- ums %>% mutate(DDEB = as.Date(DDEB, format = "%d%m%Y")
Bien sûr il est possible de réaliser l’ensemble en une seule ligne en rajoutant le %>% mutate(DDEB = as.Date(DDEB, format = "%d%m%Y") juste au bout du read_fwf() :
ums <- read_fwf( file = "DEF.UM",
col_positions = fwf_cols(UM = c(1, 4),
FINESS = c(5, 13),
AUTH = c(14, 16),
DDEB = c(17, 24),
NLITS = c(25, 27),
MODE = c(28,28)
),
col_types="ccccnc"
) %>% mutate( DDEB = as.Date(DDEB, format = "%d%m%Y"))
C’est la puissance du pipe %>% !
En lisant des fichiers ATIH contenant des prix, nous aurons souvent ce genre de modifications à faire car les formats ne représentent pas la virgule mais utilisent plutôt une mantisse fixe (2 ou 3 chiffres « après la virgule »). Il convient alors de « muter » les colonnes en les divisant respectivement par 100 ou 1000.
Conclusion
Nous avons vu aujourd’hui comment lire un fichier type du PMSI. Il suffit de décliner en fonction du format désiré.
Cette solution ne sait pas s’adapter automatiquement à la version de fichier, mais vous savez au moins comment convertir les salmigondis d’un fichier ATIH en un jeu de données correctement structuré. Bravo !